Capítulo 7 Tidy Data: uma abordagem para organizar os seus dados com tidyr
7.1 Introdução e pré-requisitos
Em qualquer análise, o formato no qual os seus dados se encontram, é muito importante. O que vamos discutir neste capítulo, será como reformatar as suas tabelas, corrigir valores não disponíveis, ou “vazios” que se encontram no formato incorreto, ou então, como preencher as suas colunas que estão incompletas de acordo com um certo padrão.
Você rapidamente descobre a importância que o formato de sua tabela carrega para o seu trabalho, na medida em que você possui pensamentos como: “Uhmm…se essa coluna estivesse na forma x, eu poderia simplesmente aplicar a função y() e todos os meus problemas estariam resolvidos”; ou então: “Se o Arnaldo não tivesse colocado os totais junto dos dados desagregados, eu não teria todo esse trabalho!”; ou talvez: “Qual é o sentido de colocar o nome dos países nas colunas? Assim fica muito mais difícil de acompanhar os meus dados!”.
Para corrigir o formato das nossas tabelas, vamos utilizar neste capítulo as funções do pacote tidyr que está incluso no tidyverse. Pelo próprio nome do pacote (tidy, que significa “arrumar”), já sabemos que ele inclui diversas funções que tem como propósito, organizar os seus dados. Portanto, lembre-se de chamar pelo pacote (seja pelo tidyr diretamente, ou pelo tidyverse) antes de prosseguir:
7.2 O que é tidy data?
Em geral, nós passamos grande parte do tempo, reorganizando os nossos dados, para que eles fiquem em um formato adequado para a nossa análise. Logo, aprender técnicas que facilitem o seu trabalho nesta atividade, pode economizar uma grande parte de seu tempo.
Isso é muito importante, pois uma base de dados que está bagunçada, é em geral bagunçada em sua própria maneira. Como resultado, cada base irá exigir um conjunto de operações e técnicas diferentes das outras bases, para que ela seja arrumada. Algumas delas, vão enfrentar problemas simples de serem resolvidos, já outras, podem estar desarrumadas em um padrão não muito bem definido, e por isso, vão dar mais trabalho para você. Por essas razões, aprender técnicas voltadas para esses problemas, se torna uma atividade necessária.
“Tidy datasets are all alike, but every messy dataset is messy in its own way”. (WICKHAM, 2014, p 2)
Toda essa problemática, ocorre não apenas pelo erro humano, mas também porque podemos representar os nossos dados de diversas maneiras em uma tabela. Sendo que essas maneiras, podem tanto facilitar muito o seu trabalho, quanto tornar o trabalho de outros, num inferno. Veja por exemplo, as tabelas abaixo. Ambas, apresentam os mesmos dados, mas em estruturas diferentes.
table2## # A tibble: 12 × 4
## country year type count
## <chr> <dbl> <chr> <dbl>
## 1 Afghanistan 1999 cases 745
## 2 Afghanistan 1999 population 19987071
## 3 Afghanistan 2000 cases 2666
## 4 Afghanistan 2000 population 20595360
## 5 Brazil 1999 cases 37737
## 6 Brazil 1999 population 172006362
## 7 Brazil 2000 cases 80488
## 8 Brazil 2000 population 174504898
## 9 China 1999 cases 212258
## 10 China 1999 population 1272915272
## 11 China 2000 cases 213766
## 12 China 2000 population 1280428583
table3## # A tibble: 6 × 3
## country year rate
## <chr> <dbl> <chr>
## 1 Afghanistan 1999 745/19987071
## 2 Afghanistan 2000 2666/20595360
## 3 Brazil 1999 37737/172006362
## 4 Brazil 2000 80488/174504898
## 5 China 1999 212258/1272915272
## 6 China 2000 213766/1280428583Antes de partirmos para a prática, vou fornecer uma base teórica que irá sustentar as suas decisões sobre como padronizar e estruturar os seus dados. Eu expliquei anteriormente, que o tidyverse é um conjunto de pacotes que dividem uma mesma filosofia. Isso significa, que esses pacotes possuem uma conexão forte entre si. Por exemplo, as funções desses pacotes, retornam os seus resultados em tibble’s, e todas as suas funções foram construídas de forma a trabalharem bem com o operador pipe (%>%). Todas essas funções também foram projetadas seguindo as melhores práticas e técnicas em análise de dados. Sendo uma dessas práticas, o que é comumente chamado na comunidade de tidy data.
O conceito de tidy data foi definido por WICKHAM (2014), e remete a forma como você está guardando os dados em sua tabela. Eu não estou dizendo aqui que todas as funções do tidyverse que apresentei até aqui, trabalham apenas com tidy data, mas sim, que essas funções são mais eficientes com essa estrutura tidy. Uma base de dados que está no formato tidy, compartilha das três seguintes características:
Cada variável de sua tabela, deve possuir a sua própria coluna.
Cada observação de sua tabela, deve possuir a sua própria linha.
Cada valor de sua tabela, deve possuir a sua própria célula.
Eu posso pressupor que essas definições acima, já são claras o suficiente para que você entenda o que são dados tidy. Porém, deixar as coisas no ar, é com certeza uma prática tão ruim quanto incluir totais junto de seus dados desagregados. Por isso, vou passar os próximos parágrafos definindo com maior precisão cada parte que compõe essas características.
Primeiro, vou definir o que quero dizer exatamente com linhas, colunas e células de sua tabela. Abaixo temos uma representação de uma base qualquer. O interesse nessa representação, não se trata dos valores e nomes inclusos nessa tabela, mas sim as áreas sombreadas dessa tabela, que estão lhe apresentando cada um dos componentes supracitados.
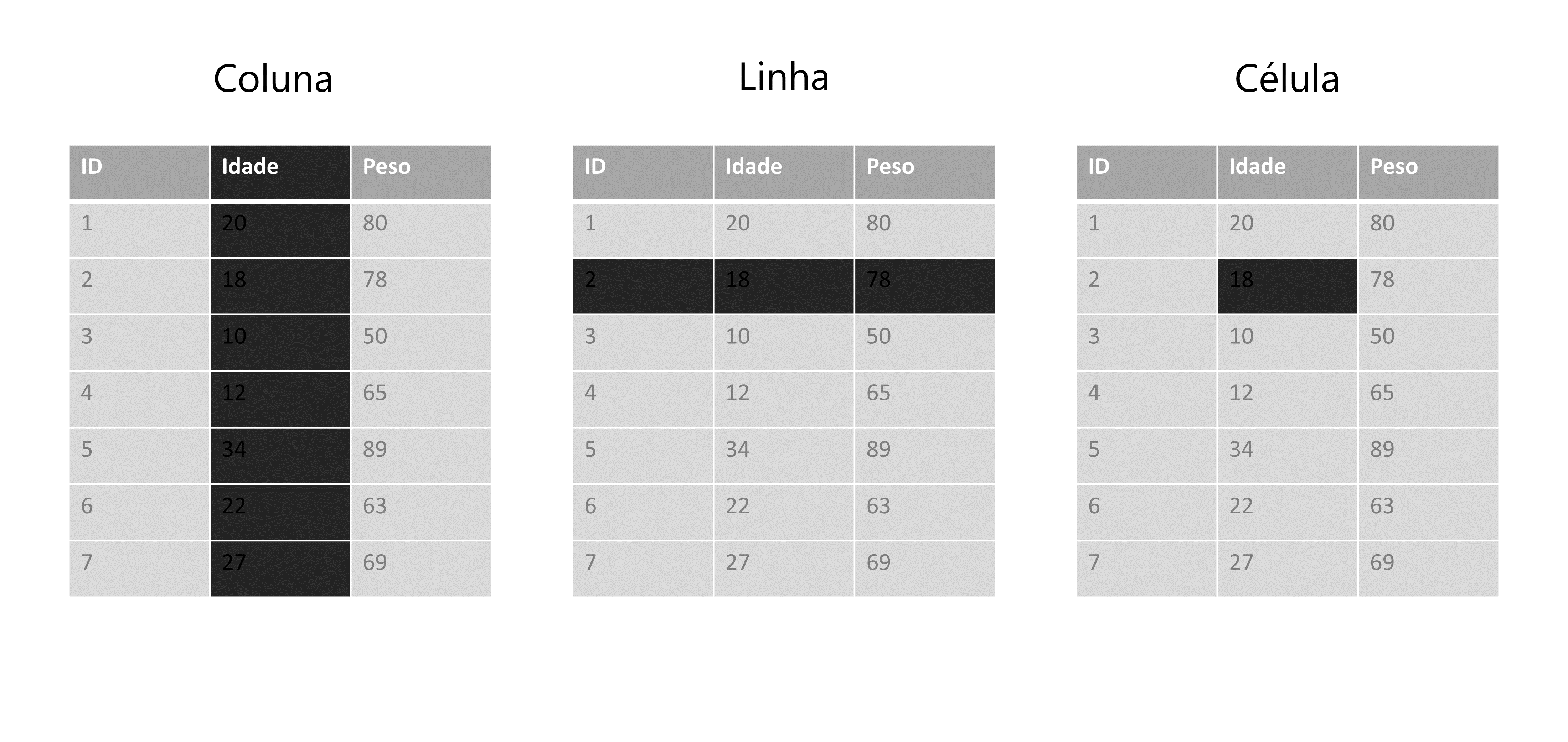
Figura 7.1: Definindo colunas, linhas e células de uma tabela
Agora, vamos definir o que são variáveis, observações e valores. Você já deve ter percebido, que toda base de dados, possui uma unidade básica que está sendo descrita ao longo dela. Ou seja, toda base lhe apresenta dados sobre um grupo específico (ou uma amostra) de algo. Esse algo pode ser um conjunto de municípios, empresas, sequências genéticas, animais, clientes, realizações de um evento estocástico, dentre outros.
Logo, se a minha base contém dados sobre os municípios do estado de Minas Gerais (MG), cada um desses municípios são uma observação de minha base. Ao dizer que cada observação deve possuir a sua própria linha, eu estou dizendo que todas as informações referentes a um município específico, devem estar em uma única linha. Em outras palavras, cada uma das 853 (total de municípios em MG) linhas da minha base, contém os dados de um município diferente do estado.
Entretanto, se a minha base descreve a evolução do PIB desses mesmos municípios nos anos de 2010 a 2020, eu não possuo mais um valor para cada município, ao longo da base. Neste momento, eu possuo 10 valores diferentes, para cada município, e mesmo que eu ainda esteja falando dos mesmos municípios, a unidade básica da minha base, se alterou. Cada um desses 10 valores, representa uma observação do PIB deste município em um ano distinto. Logo, cada um desses 10 valores para cada município, deve possuir a sua própria linha. Se o estado de Minas Gerais possui 853 municípios diferentes, isso significa que nossa base deveria ter \(10 \times 853 = 8.530\) linhas. Por isso, é importante que você preste atenção em seus dados, e identifique qual é a unidade básica que está sendo tratada.
Agora, quando eu me referir as variáveis de sua base, eu geralmente estou me referindo as colunas de sua base, porque ambos os termos são sinônimos em análises de dados. Porém, alguns cuidados são necessários, pois as variáveis de sua base podem não se encontrar nas colunas de sua tabela. Como eu disse anteriormente, há diversas formas de representar os seus dados, e por isso, há diversas formas de alocar os componentes de seus dados ao longo de sua tabela.
Uma variável de sua base de dados, não é apenas um elemento que (como o próprio nome dá a entender) varia ao longo de sua base, mas também é um elemento que lhe apresenta uma característica das suas observações. Ou seja, cada variável descreve uma característica (cor de pele, população, receita, …) de cada observação (pessoa, município, empresa, …) da minha base. O que é ou não, uma característica de sua unidade básica, irá depender de qual é essa unidade básica que está sendo descrita na base.
A população total, é uma característica geralmente associada a regiões geográficas (municípios, países, etc.), já a cor de pele pode ser uma característica de uma amostra de pessoas entrevistadas em uma pesquisa de campo (como a PNAD contínua), enquanto o número total de empresas é uma característica associada a setores da atividade econômica (CNAE - Classificação Nacional de Atividades Econômicas).
Por último, os valores de sua base, correspondem aos registros das características de cada observação de sua base. Como esse talvez seja o ponto mais claro e óbvio de todos, não vou me prolongar mais sobre ele. Pois as três características de tidy data que citamos anteriormente são interrelacionadas, de forma que você não pode satisfazer apenas duas delas. Logo, se você está satisfazendo as duas primeiras, você não precisa se preocupar com a característica que diz respeito aos valores.
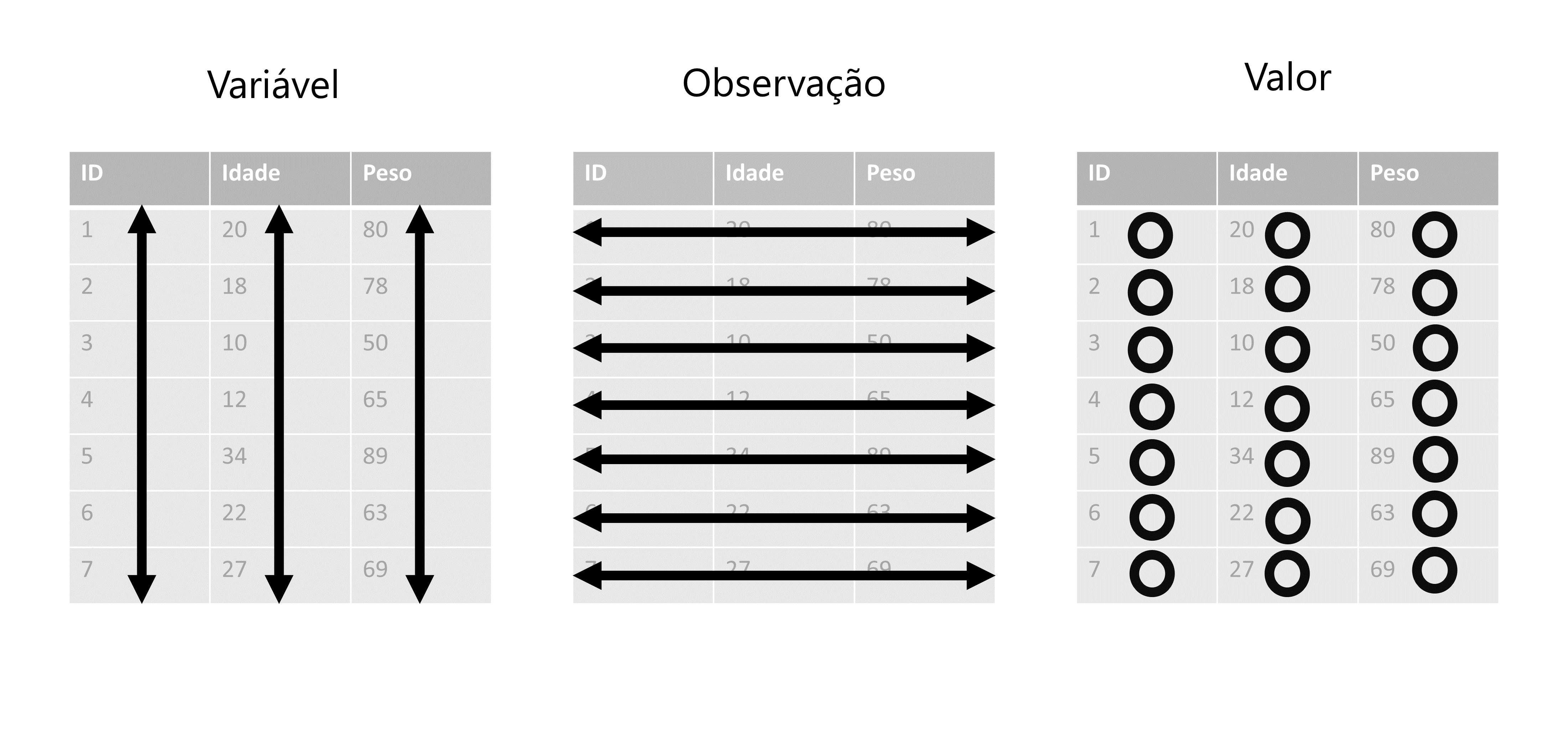
Figura 7.2: Três propriedades que caracterizam o formato tidy data
Portanto, sempre inicie o seu trabalho, identificando a unidade básica de sua base. Em seguida, tente encontrar quais são as suas variáveis, ou as características dessa unidade básica que estão sendo descritas na base. Após isso, basta alocar cada variável em uma coluna, e reservar uma linha para cada observação diferente de sua base, que você automaticamente estará deixando uma célula para cada valor da base.
7.2.1 Será que você entendeu o que é tidy data?
Nessa seção vamos fazer um teste rápido, para saber se você entendeu o que é uma tabela no formato tidy. Olhe por algum tempo para os exemplos abaixo, e reflita sobre qual dessas tabelas está no formato tidy. Tente também descobrir quais são os problemas que as tabelas “não tidy” apresentam, ou em outras palavras, qual das três definições que apresentamos anteriormente, que essas tabelas “não tidy” acabam rompendo.
table1## # A tibble: 6 × 4
## country year cases population
## <chr> <dbl> <dbl> <dbl>
## 1 Afghanistan 1999 745 19987071
## 2 Afghanistan 2000 2666 20595360
## 3 Brazil 1999 37737 172006362
## 4 Brazil 2000 80488 174504898
## 5 China 1999 212258 1272915272
## 6 China 2000 213766 1280428583
table2## # A tibble: 12 × 4
## country year type count
## <chr> <dbl> <chr> <dbl>
## 1 Afghanistan 1999 cases 745
## 2 Afghanistan 1999 population 19987071
## 3 Afghanistan 2000 cases 2666
## 4 Afghanistan 2000 population 20595360
## 5 Brazil 1999 cases 37737
## 6 Brazil 1999 population 172006362
## 7 Brazil 2000 cases 80488
## 8 Brazil 2000 population 174504898
## 9 China 1999 cases 212258
## 10 China 1999 population 1272915272
## 11 China 2000 cases 213766
## 12 China 2000 population 1280428583
table3## # A tibble: 6 × 3
## country year rate
## <chr> <dbl> <chr>
## 1 Afghanistan 1999 745/19987071
## 2 Afghanistan 2000 2666/20595360
## 3 Brazil 1999 37737/172006362
## 4 Brazil 2000 80488/174504898
## 5 China 1999 212258/1272915272
## 6 China 2000 213766/1280428583Como eu disse anteriormente, a primeira coisa que você deve fazer, é identificar a unidade básica que está sendo tratada na tabela. Nos exemplos acima, essas tabelas dizem respeito à dados de três países (Brasil, China e Afeganistão) em dois anos diferentes (1999 e 2000). Logo, a nossa tabela possui \(3 \times 2 = 6\) observações diferentes. Se uma das regras, impõe que todas as linhas devem possuir informações de uma única observação, a nossa tabela deveria possuir 6 linhas. Com isso, nós já sabemos que algo está errado com a tabela 2, pois ela possui o dobro de linhas.
Na verdade, o problema na tabela 2 é que ela está quebrando a regra de que cada variável na tabela deve possuir a sua própria coluna. Por causa dessa regra, a tabela 2 acaba extrapolando o número de linhas necessárias. Olhe para as colunas type e count. A coluna count lhe apresenta os principais valores que estamos interessados nessa tabela. Porém, a coluna type, está lhe apresentando duas variáveis diferentes.
Lembre-se de que variáveis, representam características da unidade básica de sua tabela. No nosso caso, essa unidade básica se trata dos dados anuais de países, logo, cases e population, são variáveis ou características diferentes desses países. Uma dessas variáveis está lhe apresentando um dado demográfico (população total), já a outra, está lhe trazendo um indicador epidemiológico (número de casos de alguma doença). Por isso, ambas variáveis deveriam possuir a sua própria coluna.
Ok, mas e as tabelas 1 e 3? Qual delas é a tidy? Talvez, para responder essa pergunta, você deveria primeiro procurar pela tabela “não tidy”. Veja a tabela 3, e se pergunte: “onde se encontram os valores de população e de casos de cada país nessa tabela?”. Ao se fazer essa pergunta, você provavelmente já irá descobrir qual é o problema nessa tabela.
A tabela 3, também rompe com a regra de que cada variável deve possuir a sua própria coluna. Pois o número de casos e a população total, estão guardados em uma mesma coluna! Ao separar os valores de população e de número de casos na tabela 3, em duas colunas diferentes, você chega na tabela 1, que é um exemplo de tabela tidy, pois agora todas as três definições estão sendo respeitadas.
7.2.2 Uma breve definição de formas
Apenas para que os exemplos das próximas seções, fiquem mais claros e fáceis de se visualizar mentalmente, vou definir dois formatos gerais que a sua tabela pode assumir, que são: long (longa) e wide (larga)36. Ou seja, qualquer que seja a sua tabela, ela vai em geral, estar em algum desses dois formatos, de uma forma ou de outra.
Esses termos (long e wide) são bem descritivos por si só. A ideia é que se uma tabela qualquer, está no formato long, ela adquire um aspecto visual de longa, ou em outras palavras, visualmente ela aparenta ter muitas linhas, e poucas colunas. Já uma tabela que está no formato wide, adquire um aspecto visual de larga, como se essa tabela possuísse mais colunas do que o necessário, e poucas linhas. Perceba pelos exemplos apresentados na figura 7.3, que estamos apresentando exatamente os mesmos dados, eles apenas estão organizados de formas diferentes ao longo das duas tabelas.
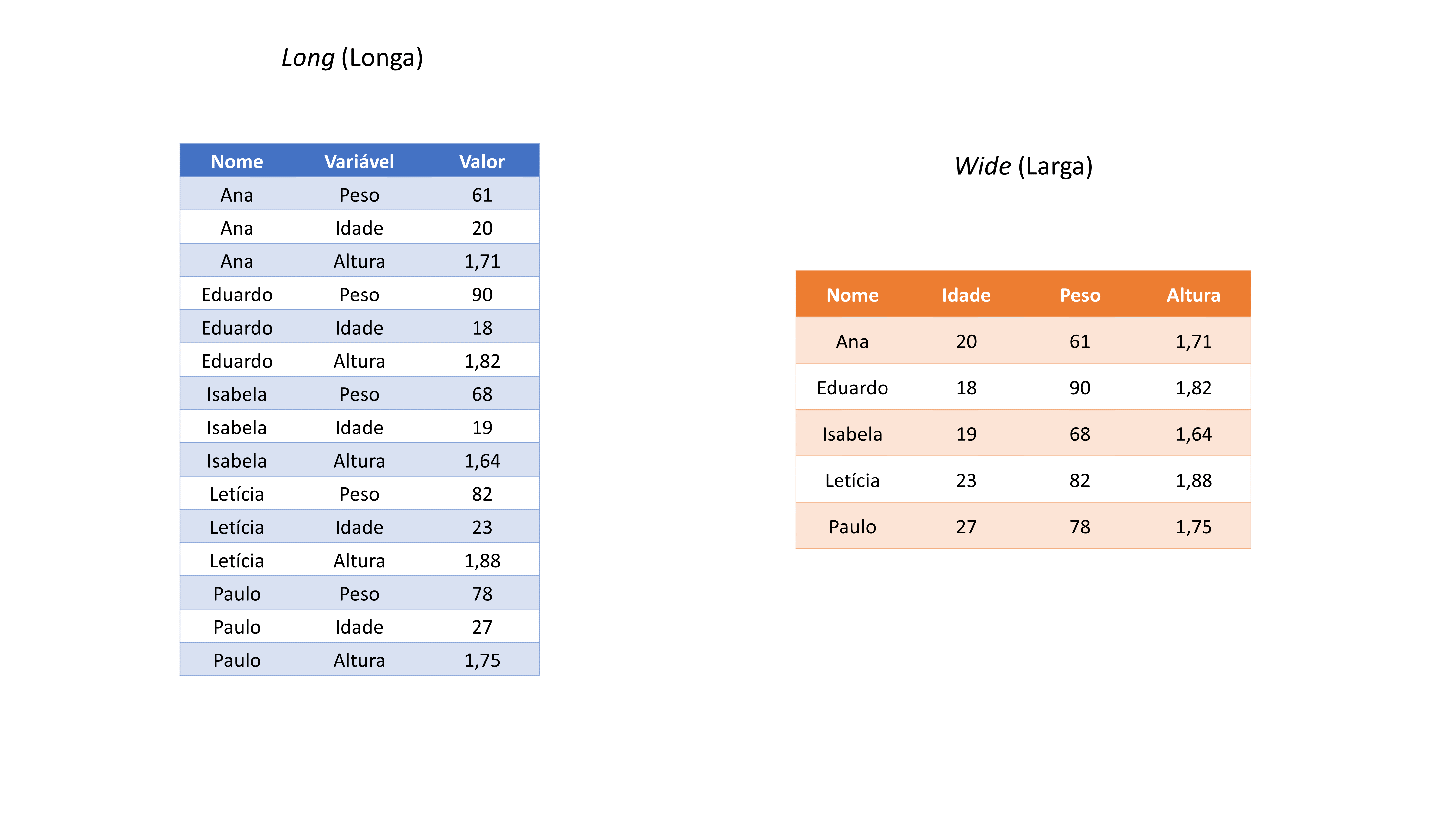
Figura 7.3: Formas gerais que a sua tabela pode adquirir
7.3 Operações de pivô
As operações de pivô são as principais operações que você irá utilizar para reformatar a sua tabela. O que essas operações fazem, é basicamente alterar as dimensões de sua tabela, ou dito de outra maneira, essas operações buscam transformar colunas em linhas, ou vice-versa. Para exemplificar essas operações, vamos utilizar as tabelas que vem do próprio pacote tidyr. Logo, se você chamou pelo tidyverse através de library(), você tem acesso a tabela abaixo. Basta chamar no console pelo objeto relig_income.
relig_income## # A tibble: 18 × 11
## religion `<$10k` `$10-20k` `$20-30k` `$30-40k` `$40-50k` `$50-75k`
## <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 Agnostic 27 34 60 81 76 137
## 2 Atheist 12 27 37 52 35 70
## 3 Buddhist 27 21 30 34 33 58
## 4 Catholic 418 617 732 670 638 1116
## 5 Don’t know/re… 15 14 15 11 10 35
## 6 Evangelical P… 575 869 1064 982 881 1486
## 7 Hindu 1 9 7 9 11 34
## 8 Historically … 228 244 236 238 197 223
## 9 Jehovah's Wit… 20 27 24 24 21 30
## 10 Jewish 19 19 25 25 30 95
## 11 Mainline Prot 289 495 619 655 651 1107
## 12 Mormon 29 40 48 51 56 112
## 13 Muslim 6 7 9 10 9 23
## 14 Orthodox 13 17 23 32 32 47
## 15 Other Christi… 9 7 11 13 13 14
## 16 Other Faiths 20 33 40 46 49 63
## 17 Other World R… 5 2 3 4 2 7
## 18 Unaffiliated 217 299 374 365 341 528
## # ℹ 4 more variables: `$75-100k` <dbl>, `$100-150k` <dbl>, `>150k` <dbl>,
## # `Don't know/refused` <dbl>Essa tabela está nos apresentando o salário médio de pessoas pertencentes a diferentes religiões. Veja que em cada coluna dessa tabela, você possui os dados de um nível (ou faixa) salarial específico. Essa é uma estrutura que pode ser fácil e intuitiva em alguns momentos, mas certamente irá trazer limites importantes para você dentro do R. Devido a especialidade que o R possui sobre operações vetorizadas, o ideal seria transformarmos essa tabela para o formato tidy.
A unidade básica dessa tabela, são os grupos religiosos, e a faixa salarial representa uma característica desses grupos. Há diferentes níveis salariais na tabela, que estão sendo distribuídos ao longo de diferentes colunas. Tendo em vista isso, uma das regras não está sendo respeita, pois todos esses diferentes níveis salarias, representam uma única característica, ou em outras palavras, eles transmitem o mesmo tipo de informação, que é um nível salarial daquele grupo religioso. Por isso, todas essas características da tabela, deve estar em uma única coluna. Em uma representação visual resumida, é isso o que precisamos fazer:
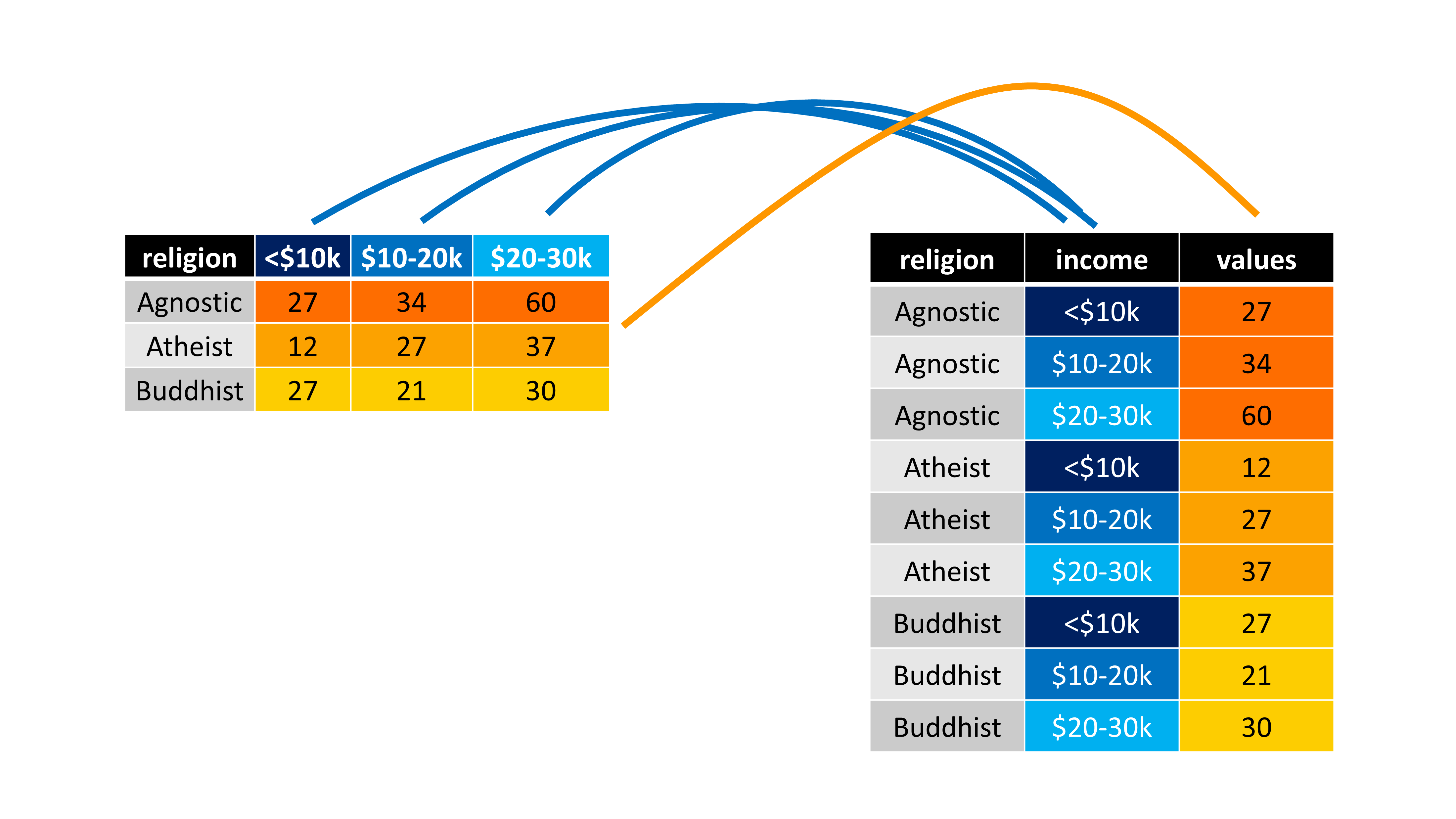
Figura 7.4: Representação de uma operação de pivô
Por isso, quando você estiver em um momento como este, em que você deseja reformatar a sua tabela, ou em outras palavras, transformar as suas linhas em colunas, ou vice-versa, você está na verdade, procurando realizar uma operação de pivô.
Nestas situações, você deve primeiro pensar como a sua tabela ficará, após a operação de pivô que você deseja aplicar. Ou seja, após essa operação, a sua tabela ficará com mais linhas/colunas? Ou menos linhas/colunas? Em outras palavras, você precisa identificar se você deseja tornar a sua tabela mais longa (aumentar o número de linhas, e reduzir o número de colunas), ou então, se você deseja torná-la mais larga (reduzir o número de linhas, e aumentar o número de colunas).
7.3.1 Adicionando linhas à sua tabela com pivot_longer()
Atualmente, a tabela relig_income possui poucas linhas e muitas colunas, e por isso, ela adquire um aspecto visual de “larga”. Como eu disse, seria muito interessante para você, que transformasse essa tabela, de modo a agrupar as diferentes faixas de níveis salarias em menos colunas. Logo, se estamos falando em reduzir o número de colunas, estamos querendo alongar a base, ou dito de outra forma, aumentar o número de linhas da base. Para fazermos isso, devemos utilizar a função pivot_longer().
Essa função possui três argumentos principais: 1) cols, os nomes das colunas que você deseja transformar em linhas; 2) names_to, o nome da nova coluna onde serão alocados os nomes, ou os rótulos das colunas que você definiu em cols; 3) values_to, o nome da nova coluna onde serão alocados os valores da sua tabela, que se encontram nas colunas que você definiu em cols. Como nós queremos transformar todas as colunas da tabela relig_income, que contém faixas salariais, eu posso simplesmente colocar no argumento cols, um símbolo de menos antes do nome da coluna religion, que é a única coluna da tabela, que não possui esse tipo de informação. Ou seja, dessa forma, eu estou dizendo à pivot_longer(), para transformar todas as colunas (exceto a coluna religion).
relig_income %>%
pivot_longer(
cols = -religion,
names_to = "income",
values_to = "values"
)## # A tibble: 180 × 3
## religion income values
## <chr> <chr> <dbl>
## 1 Agnostic <$10k 27
## 2 Agnostic $10-20k 34
## 3 Agnostic $20-30k 60
## 4 Agnostic $30-40k 81
## 5 Agnostic $40-50k 76
## 6 Agnostic $50-75k 137
## 7 Agnostic $75-100k 122
## 8 Agnostic $100-150k 109
## 9 Agnostic >150k 84
## 10 Agnostic Don't know/refused 96
## # ℹ 170 more rowsVale destacar, que você pode selecionar as colunas que você deseja transformar em linhas (argumento cols), através dos mesmos mecanismos que utilizamos na função select(). Ao eliminarmos a coluna religion com um sinal de menos (-) estávamos utilizando justamente um desses métodos. Mas podemos também, por exemplo, selecionar todas as colunas, que possuem dados de tipo numérico, com a função is.numeric(), atingindo o mesmo resultado anterior. Ou então, poderíamos selecionar todas as colunas que possuem em seu nome, algum dígito numérico, através da expressão regular "\\d" (digit) na função matches().
relig_income %>%
pivot_longer(
cols = is.numeric,
names_to = "income",
values_to = "values"
)
relig_income %>%
pivot_longer(
cols = matches("\\d"),
names_to = "income",
values_to = "values"
)Portanto, sempre que utilizar a função pivot_longer(), duas novas colunas serão criadas. Em uma dessas colunas (values_to), a função irá guardar os valores que se encontravam nas colunas que você transformou em linhas. Já na outra coluna (names_to), a função irá criar rótulos em cada linha, que lhe informam de qual coluna (que você transformou em linhas) veio o valor disposto na coluna anterior (values_to). Você sempre deve definir o nome dessas duas novas colunas, como texto, isto é, sempre forneça os nomes dessas colunas, entre aspas duplas ou simples.
Um outro exemplo, seria a tabela billboard, que também está disponível no pacote tidyr. Nessa tabela, temos a posição que diversas músicas ocuparam na lista da Billboard das 100 músicas mais populares no mundo, durante o ano de 2000. Portanto a posição que cada uma dessas músicas ocuparam nessa lista, ao longo do tempo, é a unidade básica que está sendo tratada nessa tabela. Agora, repare que a tabela possui muitas colunas (79 no total), onde em cada uma delas, temos a posição de uma música em uma dada semana desde a sua entrada na lista.
billboard## # A tibble: 317 × 79
## artist track date.entered wk1 wk2 wk3 wk4 wk5 wk6 wk7
## <chr> <chr> <date> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 2 Pac Baby… 2000-02-26 87 82 72 77 87 94 99
## 2 2Ge+her The … 2000-09-02 91 87 92 NA NA NA NA
## 3 3 Doors Do… Kryp… 2000-04-08 81 70 68 67 66 57 54
## 4 3 Doors Do… Loser 2000-10-21 76 76 72 69 67 65 55
## 5 504 Boyz Wobb… 2000-04-15 57 34 25 17 17 31 36
## 6 98^0 Give… 2000-08-19 51 39 34 26 26 19 2
## 7 A*Teens Danc… 2000-07-08 97 97 96 95 100 NA NA
## 8 Aaliyah I Do… 2000-01-29 84 62 51 41 38 35 35
## 9 Aaliyah Try … 2000-03-18 59 53 38 28 21 18 16
## 10 Adams, Yol… Open… 2000-08-26 76 76 74 69 68 67 61
## # ℹ 307 more rows
## # ℹ 69 more variables: wk8 <dbl>, wk9 <dbl>, wk10 <dbl>, wk11 <dbl>,
## # wk12 <dbl>, wk13 <dbl>, wk14 <dbl>, wk15 <dbl>, wk16 <dbl>,
## # wk17 <dbl>, wk18 <dbl>, wk19 <dbl>, wk20 <dbl>, wk21 <dbl>,
## # wk22 <dbl>, wk23 <dbl>, wk24 <dbl>, wk25 <dbl>, wk26 <dbl>,
## # wk27 <dbl>, wk28 <dbl>, wk29 <dbl>, wk30 <dbl>, wk31 <dbl>,
## # wk32 <dbl>, wk33 <dbl>, wk34 <dbl>, wk35 <dbl>, wk36 <dbl>, …Repare também, que temos nessa tabela, mais semanas do que o total de semanas contidas em um ano corrido (\(365/7 \approx 52\) semanas). Pela descrição das colunas restantes, que se encontra logo abaixo da tabela, vemos que a tabela possui dados até a 76° semana (wk76). Isso provavelmente ocorre, porque algumas músicas que estão sendo descritas nessa tabela, entraram para a lista da Billboard no meio do ano anterior (1999), e, portanto, permaneceram na lista mesmo durante o ano de 2000, ultrapassando o período de 1 ano, e, portanto, de 52 semanas.
Agora, está claro que a forma como essa tabela está organizada, pode lhe trazer um trabalho imenso. Especialmente se você precisar aplicar uma função sobre cada uma dessas 76 colunas separadamente. Por isso, o ideal seria transformarmos todas essas 76 colunas, em novas linhas de sua tabela.
Porém, você não vai querer digitar o nome de cada uma dessas 76 colunas, no argumento cols de pivot_longer(). Novamente, quando há um conjunto muito grande de colunas que desejamos selecionar, podemos utilizar os métodos alternativos de seleção que vimos em select(). Por exemplo, podemos selecionar todas essas colunas pelos seus índices. No primeiro exemplo abaixo, estamos fazendo justamente isso, ao dizer à função em cols, que desejamos transformar todas as colunas entre a 4° e a 79° coluna. Uma outra alternativa, seria selecionarmos todas as colunas que possuem nomes que começam por “wk”, com a função starts_with(). Ambas alternativas, geram o mesmo resultado.
billboard_long <- billboard %>%
pivot_longer(
cols = 4:79,
names_to = "week",
values_to = "position"
)
billboard_long <- billboard %>%
pivot_longer(
cols = starts_with("wk"),
names_to = "week",
values_to = "position"
)
billboard_long## # A tibble: 24,092 × 5
## artist track date.entered week position
## <chr> <chr> <date> <chr> <dbl>
## 1 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk1 87
## 2 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk2 82
## 3 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk3 72
## 4 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk4 77
## 5 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk5 87
## 6 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk6 94
## 7 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk7 99
## 8 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk8 NA
## 9 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk9 NA
## 10 2 Pac Baby Don't Cry (Keep... 2000-02-26 wk10 NA
## # ℹ 24,082 more rowsTais métodos de seleção são muito eficazes, e trazem grande otimização para o seu trabalho. Entretanto, em muitas ocasiões que utilizar essas funções de pivô, você vai precisar transformar apenas um conjunto pequeno de colunas em sua tabela. Nestes casos, talvez seja mais simples, definir diretamente os nomes das colunas que você deseja transformar, em cols. Veja por exemplo, a tabela df que eu crio logo abaixo.
df <- tibble(
nome = c("Ana", "Eduardo", "Paulo"),
`2005` = c(1800, 2100, 1230),
`2006` = c(2120, 2100, 1450),
`2007` = c(2120, 2100, 1980),
`2008` = c(3840, 2100, 2430)
)
df## # A tibble: 3 × 5
## nome `2005` `2006` `2007` `2008`
## <chr> <dbl> <dbl> <dbl> <dbl>
## 1 Ana 1800 2120 2120 3840
## 2 Eduardo 2100 2100 2100 2100
## 3 Paulo 1230 1450 1980 2430Essa tabela contém os salários médios de três indivíduos hipotéticos, ao longo de quatro anos diferentes. Note que esses quatro anos, estão distribuídos ao longo de quatro colunas dessa tabela. Nesse exemplo, podemos utilizar novamente a função pivot_longer(), para transformarmos essas colunas em linhas. Dessa forma, temos o seguinte resultado:
df %>%
pivot_longer(
cols = c("2005", "2006", "2007", "2008"),
names_to = "ano",
values_to = "salario"
)## # A tibble: 12 × 3
## nome ano salario
## <chr> <chr> <dbl>
## 1 Ana 2005 1800
## 2 Ana 2006 2120
## 3 Ana 2007 2120
## 4 Ana 2008 3840
## 5 Eduardo 2005 2100
## 6 Eduardo 2006 2100
## 7 Eduardo 2007 2100
## 8 Eduardo 2008 2100
## 9 Paulo 2005 1230
## 10 Paulo 2006 1450
## 11 Paulo 2007 1980
## 12 Paulo 2008 2430
7.3.2 Adicionando colunas à sua tabela com pivot_wider()
Por outro lado, você talvez deseje realizar a operação contrária. Ou seja, se você deseja transformar linhas de sua tabela, em novas colunas, você deve utilizar a função pivot_wider(), que possui argumentos muito parecidos com os de pivot_longer().
Vamos começar com um exemplo simples. Veja a tabela df que estou criando logo abaixo. Nessa tabela, temos dados como o peso, a idade e a altura de cinco pessoas diferentes. Porém, perceba que essa tabela, não está no formato tidy. Pois temos três informações (peso, idade e altura) que representam características diferentes da unidade básica da tabela (pessoas), que estão em uma mesma coluna (variavel).
df <- structure(list(nome = c("Ana", "Ana", "Ana", "Eduardo", "Eduardo",
"Eduardo", "Paulo", "Paulo", "Paulo", "Henrique", "Henrique",
"Henrique", "Letícia", "Letícia", "Letícia"), variavel = c("idade",
"peso", "altura", "idade", "peso", "altura", "idade", "peso",
"altura", "idade", "peso", "altura", "idade", "peso", "altura"
), valor = c(20, 61, 1.67, 18, 90, 1.89, 19, 68, 1.67, 23, 82,
1.72, 27, 56, 1.58)), row.names = c(NA, -15L), class = c("tbl_df",
"tbl", "data.frame"))
df## # A tibble: 15 × 3
## nome variavel valor
## <chr> <chr> <dbl>
## 1 Ana idade 20
## 2 Ana peso 61
## 3 Ana altura 1.67
## 4 Eduardo idade 18
## 5 Eduardo peso 90
## 6 Eduardo altura 1.89
## 7 Paulo idade 19
## 8 Paulo peso 68
## 9 Paulo altura 1.67
## 10 Henrique idade 23
## 11 Henrique peso 82
## 12 Henrique altura 1.72
## 13 Letícia idade 27
## 14 Letícia peso 56
## 15 Letícia altura 1.58Portanto, tendo identificado o problema, precisamos agora, separar as três variáveis contidas na coluna variavel, em três novas colunas da tabela df. Logo, precisamos alargar a nossa base, pois estamos eliminando linhas e adicionando colunas à tabela.
Já sabemos que podemos utilizar a função pivot_wider() para esse trabalho, mas eu ainda não descrevi os seus argumentos, que são os seguintes: 1) id_cols, sendo as colunas que são suficientes para, ou capazes de, identificar uma única observação de sua base; 2) names_from, qual a coluna de sua tabela que contém as linhas a serem dividas, ou transformadas, em várias outras colunas; 3) values_from, qual a coluna, que contém os valores a serem posicionados nas novas células, que serão criadas durante o processo de “alargamento” da sua tabela.
Antes de prosseguirmos para os exemplos práticos, é provavelmente uma boa ideia, refletirmos sobre o que o argumento id_cols significa. Para que você identifique as colunas a serem estipuladas no argumento id_cols, você precisa primeiro identificar a unidade básica que está sendo tratada em sua tabela. No nosso caso, a tabela df, contém dados sobre características físicas ou biológicas, de cinco pessoas diferentes. Logo, a unidade básica dessa tabela, são as pessoas que estão sendo descritas nela, e por isso, a coluna nome é capaz de identificar cada unidade básica, pois ela nos traz justamente um código social de identificação, isto é, o nome dessas pessoas.
Porém, repare que cada pessoa descrita na tabela df, não possui a sua própria linha na tabela. Veja por exemplo, as informações referentes à Ana, que estão definidas ao longo das três primeiras linhas da tabela. Com isso, eu quero apenas destacar que cada unidade básica, ou cada observação de sua tabela, não necessariamente vai se encontrar em uma única linha, e que isso não deve ser uma regra (ou um guia) para selecionarmos as colunas de id_cols. Até porque, nós estamos utilizando uma operação de pivô sobre a nossa tabela, justamente pelo fato dela não estar no formato tidy. Ou seja, se uma das características que definem o formato tidy, não estão sendo respeitados, é muito provável, que cada observação de sua base, não se encontre em uma única linha.
Pensando em um outro exemplo, se você dispõe de uma base que descreve o PIB de cada município do estado de Minas Gerais, você precisa definir em id_cols, a coluna (ou o conjunto de colunas) que é capaz de identificar cada um dos 853 municípios de MG, pois esses municípios são a unidade básica da tabela. Porém, se a sua base está descrevendo o PIB desses mesmos municípios, mas agora ao longo dos anos de 2010 a 2020, a sua unidade básica passa a ter um componente temporal, e se torna a evolução desses municípios ao longo do tempo. Dessa forma, você precisaria não apenas de uma coluna que seja capaz de identificar qual o município que está sendo descrito na base, mas também de uma outra coluna que possa identificar qual o ano que a informação desse município se refere.
Tendo isso em mente, vamos partir para os próximos dois argumentos. No nosso caso, queremos pegar as três variáveis que estão ao longo da coluna variavel, e separá-las em três colunas diferentes. Isso é exatamente o que devemos definir em names_from. O que este argumento está pedindo, é o nome da coluna que contém os valores que vão servir de nome para as novas colunas que pivot_wider() irá criar. Ou seja, ao fornecermos a coluna variavel para names_from, pivot_wider() irá criar uma nova coluna para cada valor único que se encontra na coluna variavel. Como ao longo da coluna variavel, temos três valores diferentes (peso, altura e idade), pivot_wider() irá criar três novas colunas que possuem os nomes de peso, altura e idade.
Ao criar as novas colunas, você precisa preenchê-las de alguma forma, a menos que você deseja deixá-las vazias. Em outras palavras, a função pivot_wider() irá lhe perguntar: “Ok, eu criei as colunas que você me pediu para criar, mas eu devo preenchê-las com que valores?”. Você deve responder essa pergunta, através do argumento values_from, onde você irá definir qual é a coluna que contém os valores que você deseja alocar ao longo dessas novas colunas (que foram criadas de acordo com os valores contidos na coluna que você definiu em names_from). Na nossa tabela df, é a coluna valor que contém os registros, ou os valores que cada variável (idade, altura e peso) assume nessa amostra. Logo, é essa coluna que devemos conectar à values_from.
df %>%
pivot_wider(
id_cols = nome,
names_from = variavel,
values_from = valor
)## # A tibble: 5 × 4
## nome idade peso altura
## <chr> <dbl> <dbl> <dbl>
## 1 Ana 20 61 1.67
## 2 Eduardo 18 90 1.89
## 3 Paulo 19 68 1.67
## 4 Henrique 23 82 1.72
## 5 Letícia 27 56 1.58Esse foi um exemplo simples de como utilizar a função, e que vai lhe servir de base para praticamente qualquer aplicação de pivot_wider(). Porém, em algumas situações que você utilizar pivot_wider(), pode ser que a sua tabela não possua colunas o suficiente, que possam identificar unicamente cada observação de sua base, e isso, ficará mais claro com um outro exemplo.
Com o código abaixo, você é capaz de recriar a tabela vendas, em seu R. Lembre-se de executar a função set.seed() antes de criar a tabela vendas, pois é essa função que garante que você irá recriar exatamente a mesma tabela que a minha. Nessa tabela vendas, possuímos vendas hipóteticas de diversos produtos (identificados por produtoid), realizadas por alguns vendedores (identificados por usuario) que arrecadaram em cada venda os valores descritos na coluna valor. Perceba também, que essas vendas são diárias, pois possuímos outras três colunas (ano, mes e dia) que definem o dia em que a venda ocorreu.
nomes <- c("Ana", "Eduardo", "Paulo", "Henrique", "Letícia")
produto <- c("10032", "10013", "10104", "10555", "10901")
set.seed(1)
vendas <- tibble(
ano = sample(2010:2020, size = 10000, replace = TRUE),
mes = sample(1:12, size = 10000, replace = TRUE),
dia = sample(1:31, size = 10000, replace = TRUE),
usuario = sample(nomes, size = 10000, replace = TRUE),
valor = rnorm(10000, mean = 5000, sd = 1600),
produtoid = sample(produto, size = 10000, replace = TRUE)
) %>%
arrange(ano, mes, dia, usuario)
vendas## # A tibble: 10,000 × 6
## ano mes dia usuario valor produtoid
## <int> <int> <int> <chr> <dbl> <chr>
## 1 2010 1 1 Ana 3907. 10104
## 2 2010 1 1 Henrique 6139. 10104
## 3 2010 1 2 Henrique 5510. 10013
## 4 2010 1 3 Ana 5296. 10555
## 5 2010 1 3 Letícia 3525. 10555
## 6 2010 1 4 Ana 5102. 10555
## 7 2010 1 4 Eduardo 6051. 10013
## 8 2010 1 4 Letícia 4600. 10032
## 9 2010 1 5 Paulo 5869. 10104
## 10 2010 1 6 Ana 7188. 10013
## # ℹ 9,990 more rowsVou antes de mais nada, identificar os níveis (ou valores únicos) contidos nas duas colunas que vão servir de objeto de estudo, para os próximos exemplos. Caso você queira visualizar todos os valores únicos contidos em uma coluna, você pode realizar tal ação através da função unique(). Perceba pelos resultados abaixo, que nós temos cinco vendedores e cinco produtos diferentes que estão sendo descritos ao longo da tabela vendas.
unique(vendas$usuario)## [1] "Ana" "Henrique" "Letícia" "Eduardo" "Paulo"
unique(vendas$produtoid)## [1] "10104" "10013" "10555" "10032" "10901"Portanto, vamos para o exemplo. Já adianto, que se você tentar distribuir tanto os vendedores (usuario), quanto os produtos vendidos (produtoid), em novas colunas de nossa tabela, utilizando pivot_wider(), um aviso será levantado, e o resultado dessa operação (apesar de correto) será provavelmente, muito estranho para você. Primeiro, veja com os seus próprios olhos, qual é o resultado dessa aplicação, com a coluna usuario:
vendas_wide <- vendas %>%
pivot_wider(
id_cols = c("ano", "mes", "dia", "produtoid"),
names_from = usuario,
values_from = valor
)## Warning: Values from `valor` are not uniquely identified; output will contain
## list-cols.
## • Use `values_fn = list` to suppress this warning.
## • Use `values_fn = {summary_fun}` to summarise duplicates.
## • Use the following dplyr code to identify duplicates.
## {data} %>%
## dplyr::group_by(ano, mes, dia, produtoid, usuario) %>%
## dplyr::summarise(n = dplyr::n(), .groups = "drop") %>%
## dplyr::filter(n > 1L)
vendas_wide## # A tibble: 7,845 × 9
## ano mes dia produtoid Ana Henrique Letícia Eduardo Paulo
## <int> <int> <int> <chr> <list> <list> <list> <list> <list>
## 1 2010 1 1 10104 <dbl [1]> <dbl [1]> <NULL> <NULL> <NULL>
## 2 2010 1 2 10013 <NULL> <dbl [1]> <NULL> <NULL> <NULL>
## 3 2010 1 3 10555 <dbl [1]> <NULL> <dbl [1]> <NULL> <NULL>
## 4 2010 1 4 10555 <dbl [1]> <NULL> <NULL> <NULL> <NULL>
## 5 2010 1 4 10013 <NULL> <NULL> <NULL> <dbl> <NULL>
## 6 2010 1 4 10032 <NULL> <NULL> <dbl [1]> <NULL> <NULL>
## 7 2010 1 5 10104 <NULL> <NULL> <NULL> <NULL> <dbl>
## 8 2010 1 6 10013 <dbl [1]> <NULL> <NULL> <NULL> <NULL>
## 9 2010 1 6 10901 <NULL> <NULL> <dbl [1]> <NULL> <NULL>
## 10 2010 1 7 10013 <NULL> <dbl [1]> <NULL> <NULL> <dbl>
## # ℹ 7,835 more rowsComo podemos ver pelo resultado acima, uma mensagem de aviso apareceu, nos informando que os valores não podem ser unicamente identificados através das colunas que fornecemos em id_cols (Values are not uniquely identified), e que por isso, a função pivot_wider(), acabou transformando as novas colunas que criamos, em listas (output will contain list-cols.).
Ou seja, cada uma das colunas que acabamos de criar com pivot_wider(), estão na estrutura de um vetor recursivo (i.e. listas). Isso pode ser estranho para muitos usuários, pois na maioria das vezes, as colunas de suas tabelas serão vetores atômicos37. Um outro motivo que provavelmente levantou bastante dúvida em sua cabeça é: “Como assim as colunas que forneci não são capazes de identificar unicamente os valores? Em que sentido elas não são capazes de realizar tal ação?”. Bem, essa questão ficará mais clara, se nos questionarmos como, ou por que motivo essas colunas foram transformadas para listas.
Antes de continuarmos, vale ressaltar que as novas colunas criadas por pivot_wider() nunca chegaram a ser colunas comuns, formadas por vetores atômicos. Logo, desde a sua criação, elas já eram listas. Mas se partirmos do pressuposto que inicialmente, essas colunas eram vetores atômicos, tal pensamento se torna útil para identificarmos os motivos para o uso de listas. Estes motivos serão identificados a seguir.
Primeiro, precisamos transformar novamente essas colunas em vetores atômicos, para tentarmos compreender como essas colunas ficariam como simples vetores atômicos. Para isso, vou pegar um pedaço da tabela vendas, mais especificamente, as 10 primeiras linhas da tabela, através da função head(). Em seguida, vou me preocupar em transformar essas colunas novamente em vetores, através dos comandos abaixo.
pedaco <- head(vendas_wide, 10)
for(i in 5:9){
id <- vapply(pedaco[[i]], FUN = is.null, FUN.VALUE = TRUE)
pedaco[[i]][id] <- NA_real_
}
pedaco <- pedaco %>% mutate(across(5:9, unlist))Após executarmos as transformações acima, possuímos agora, uma tabela comum, como qualquer outra que você encontra normalmente no R. Veja o resultado abaixo, quando chamamos pelo nome da tabela no console. Dessa vez, nas células que possuíam uma lista nula <NULL>(uma lista vazia) temos um valor de NA (não disponível). Já nas células que possuíam uma lista com algum valor, vemos agora, o valor exato que estava contido nessa lista, ao invés da descrição <dbl [1]>.
pedaco## # A tibble: 10 × 9
## ano mes dia produtoid Ana Henrique Letícia Eduardo Paulo
## <int> <int> <int> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 2010 1 1 10104 3907. 6139. NA NA NA
## 2 2010 1 2 10013 NA 5510. NA NA NA
## 3 2010 1 3 10555 5296. NA 3525. NA NA
## 4 2010 1 4 10555 5102. NA NA NA NA
## 5 2010 1 4 10013 NA NA NA 6051. NA
## 6 2010 1 4 10032 NA NA 4600. NA NA
## 7 2010 1 5 10104 NA NA NA NA 5869.
## 8 2010 1 6 10013 7188. NA NA NA NA
## 9 2010 1 6 10901 NA NA 4491. NA NA
## 10 2010 1 7 10013 NA 4407. NA NA 2292.Portanto, se observarmos a primeira linha dessa nova tabela pedaco, vemos que a vendedora Ana, vendeu no dia 01/01/2010, o produto de ID 10104, no valor de 3907 reais (e alguns centavos). Neste mesmo dia, o Henrique vendeu o mesmo produto, por quase 2 mil reais a mais que Ana, totalizando 6139 reais de receita. Podemos perceber também pelas outras colunas, que nenhum outro vendedor conseguiu vender uma unidade do produto de ID 10104, no dia 01/01/2010.
Neste ponto, se pergunte: “Ok, Ana vendeu uma unidade do produto 10104, no dia 01. Mas e se ela tivesse vendido duas unidades desse mesmo produto 10104, no dia 01?”. Tente imaginar, como os dados dessas duas vendas ficariam na tabela. Isso não é uma questão trivial, pois temos agora dados de duas vendas diferentes…mas apenas uma célula disponível em nossa tabela, para guardar esses dados. É a partir deste choque, que podemos identificar qual foi o motivo para o uso de listas nas novas colunas.
Dito de outra forma, temos uma única célula na tabela (localizada na primeira linha, e quinta coluna da tabela), que deve conter o valor arrecadado na venda do produto 10104, realizada pela vendedora Ana no dia 01/01/2010. Você poderia pensar: “Bem, por que não somar os valores dessas duas vendas? Dessa forma, temos apenas um valor para encaixar nessa célula”. Essa é uma alternativa possível, porém, ela gera perda de informação, especialmente se o valor arrecadado nas duas operações forem diferentes. Por exemplo, se a receita da primeira venda foi de 3907, e da segunda, de 4530. Com essa alternativa, nós sabemos que a soma das duas vendas ocorridas naquele dia, geraram 8437 reais de receita, mas nós não sabemos mais, qual foi o menor valor arrecadado nas duas operações.
Isso é particularmente importante, pois podemos gerar o mesmo valor (8437 reais) de múltiplas formas. Pode ser que a Ana tenha vendido cinco unidades do produto 10104, tendo arrecadado em cada venda, o valor de 1687,4 reais. Mas ela poderia atingir o mesmo valor, ao vender dez unidades do produto 10104, dessa vez, arrecadando um valor médio bem menor, de 843,7 reais. Portanto, se utilizarmos a soma desses valores, como forma de contornarmos o problema posto anteriormente, os administradores da loja não poderão mais inferir da tabela vendas, se as suas vendas tem se reduzido em quantidade, ou se o valor arrecadado em cada venda, tem caído ao longo dos últimos anos.
Apesar de ser uma alternativa ruim para muitos casos, pode ser desejável agregar as informações dessas vendas em uma só para o seu caso. Nesta situação, as versões mais recentes do pacote tidyr, oferecem na função pivot_wider() o argumento values_fn, onde você pode fornecer o nome de uma função a ser aplicada sobre os valores dispostos em cada célula. Logo, se quiséssemos somar os valores de vendas de cada célula criada na tabela vendas_wide, poderíamos realizar os comandos abaixo:
vendas %>%
pivot_wider(
id_cols = c("ano", "mes", "dia", "produtoid"),
names_from = usuario,
values_from = valor,
values_fn = sum
)## # A tibble: 7,845 × 9
## ano mes dia produtoid Ana Henrique Letícia Eduardo Paulo
## <int> <int> <int> <chr> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 2010 1 1 10104 3907. 6139. NA NA NA
## 2 2010 1 2 10013 NA 5510. NA NA NA
## 3 2010 1 3 10555 5296. NA 3525. NA NA
## 4 2010 1 4 10555 5102. NA NA NA NA
## 5 2010 1 4 10013 NA NA NA 6051. NA
## 6 2010 1 4 10032 NA NA 4600. NA NA
## 7 2010 1 5 10104 NA NA NA NA 5869.
## 8 2010 1 6 10013 7188. NA NA NA NA
## 9 2010 1 6 10901 NA NA 4491. NA NA
## 10 2010 1 7 10013 NA 4407. NA NA 2292.
## # ℹ 7,835 more rowsRecapitulando, nossa hipótese, é de que tenha ocorrido mais de uma venda de um mesmo produto, por um mesmo vendedor, em um mesmo dia na tabela vendas. Para comprovar se essa hipótese ocorre ou não em nossa tabela, podemos coletar o número de observações contidas em cada célula da coluna Ana, por exemplo, e verificarmos se há algum valor acima de 1. Vale ressaltar que as colunas criadas por pivot_wider() em vendas_wide, são agora listas, e principalmente, que desejamos coletar o número de observações contidas em cada um dos elementos da lista que representa a coluna Ana. Para isso, precisamos de algo como os comandos abaixo:
vec <- vector(mode = "double", length = nrow(vendas_wide))
for(i in seq_along(vendas_wide$Ana)){
vec[i] <- length(vendas_wide$Ana[[i]])
}
vec[vec > 1]## [1] 3 2 2 2 2 2 2 2 2 2 2 2 2 3 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2
## [36] 2 2 2 2 2 2 2 2 3 2 2 2 2 2 2 2 2 2 2 2 3 3 2 2 2 2 2 2 2 2 2 2 2 2 3
## [71] 3 2 2 2 2 2 2 2 2 2 2 3 2 2 2 2 2 2 2 2 2 2 3 3 2 2 2Podemos ver pelo resultado acima, que sim, possuímos dias em que a vendedora Ana, vendeu mais de uma vez, o mesmo produto. É neste sentido que a função pivot_wider() gerou aquele aviso para nós. A função estava nos informando que ela não possuía meios de identificar cada venda realizada pela vendedora Ana desse mesmo produto, nesses dias que foram provavelmente movimentados na loja. Nós fornecemos ao argumento id_cols, as colunas ano, mes, dia e produtoid. Porém, essas colunas em conjunto não são capazes de diferenciar as três vendas de Ana do produto 10013 que ocorreram no dia 29/02/2010, por exemplo, nem as duas vendas de Henrique do produto 10104 no dia 18/01/2010, e muitas outras. Foi por esse motivo, que listas foram utilizadas nas novas colunas de pivot_wider().
Você talvez pense: “Por que não fornecemos então todas as colunas da tabela para id_cols?”. Primeiro, esse questionamento carrega um pressuposto que não necessariamente se confirma, que é o de que os valores das vendas realizadas por um mesmo vendedor, de um mesmo produto, e no mesmo dia, são diferentes em todas as ocasiões. Algo que é possível, mas não necessariamente ocorre ao longo de toda a base. O segundo problema, é que a coluna valor não está mais disponível para ser utilizada por id_cols, pois se você se lembrar, nós conectamos essa coluna a values_from. Isso significa, que essa coluna já está sendo utilizada para preencher as novas células que estão sendo criadas por pivot_wider(), e, portanto, ela não pode ocupar dois espaços ao mesmo tempo. Tanto que se você tentar adicionar a coluna valor a id_cols, você irá perceber que nada se altera, e o mesmo resultado é gerado.
Portanto, não há uma resposta fácil para uma situação como essa, onde mesmo fornecendo todas as colunas para id_cols em pivot_wider(), a função ainda não é capaz de identificar unicamente cada valor da coluna que você forneceu em value_from. Você pode utilizar uma solução que gera perda de informação, ao aplicar uma função sumária, ou seja, uma função para agregar esses valores de forma que eles se tornem únicos, dados os conjuntos de colunas que você forneceu em id_cols. Uma outra possibilidade, é que você esteja utilizando a operação de pivô errada. Ou seja, a melhor alternativa seria alongar (pivot_longer()) a sua base, ao invés de alargá-la.
Agora, uma última possibilidade mais promissora, é que você esteja realizando a operação correta, e que faz sentido manter essas colunas como listas de acordo com o que você deseja realizar com a base. Isso inclui o uso de um ferramental que está um pouco além desse capítulo. Por outro lado, lidar com nested data, é mais uma questão de experiência, de se acostumar com tal estrutura, e saber as funções adequadas, do que aprender algo muito diferente do que mostramos aqui. Um outro conhecimento que é de extrema importância nessas situações, é conhecer muito bem como as listas funcionam no R. Se você conhecer bem essa estrutura, você não terá dificuldades em navegar por nested data. Para uma visão melhor do potencial que nested data pode trazer para sua análise, eu recomendo que você procure por uma excelente palestra de Hadley Wickham, entitulada “Managing many models with R”38.
7.4 Completando e expandindo a sua tabela
A operação que vou mostrar a seguir, serve para completar, ou inserir linhas que estão faltando em sua tabela. Em outras palavras, essa operação busca tornar os valores que estão implicitamente faltando em sua tabela, em valores não disponíveis explícitos. Você também pode enxergar esse processo, como uma forma rápida de expandir a sua tabela, a partir de combinações de valores. Um exemplo lógico do uso dessa operação, seriam datas que você gostaria que estivessem em sua tabela, mas que não se encontram nela no momento. Vamos supor por exemplo, que você possua a tabela abaixo:
library(tidyverse)
dias <- c("2020-09-01", "2020-09-05", "2020-09-07", "2020-09-10")
set.seed(1)
vendas <- tibble(
datas = as.Date(dias),
nome = c("Ana", "Julia", "Joao", "Julia"),
valor = rnorm(4, mean = 500, sd = 150)
)
vendas## # A tibble: 4 × 3
## datas nome valor
## <date> <chr> <dbl>
## 1 2020-09-01 Ana 406.
## 2 2020-09-05 Julia 528.
## 3 2020-09-07 Joao 375.
## 4 2020-09-10 Julia 739.Portanto, temos nessa tabela vendas, o nome de alguns vendedores e os valores de suas vendas efetuadas em alguns dias diferentes. No momento, temos vendas explicitas apenas nos dias 01, 05, 07 e 10 de setembro de 2020, mas o que ocorreu nos dias que estão entre essas datas (dias 02, 03, 04, 06, 08 e 09 de setembro de 2020)? Caso você estivesse apresentando esses dados para o seu chefe, por exemplo, essa seria uma questão que ele provavelmente faria a você.
Bem, vamos supor que não tenham ocorrido vendas durante esses dias, e que por isso eles não estão sendo descritos na tabela vendas. Talvez seja de seu desejo, introduzir esses dias na tabela para que ninguém fique em dúvida a respeito desses dias. Com isso, precisamos então completar a tabela vendas, com linhas que estão implicitamente faltando nela.
7.4.1 Encontrando possíveis combinações com a função expand()
Apesar não ser exatamente o que desejamos para a tabela vendas, o processo em que buscamos encontrar possíveis combinações de dados que não estão presentes em nossa tabela, também envolve a procura por todas as combinações possíveis dos dados presentes nessa tabela. Nessa seção, vamos introduzir alguns métodos para encontrarmos todas as combinações possíveis de seus dados.
Para isso, podemos utilizar a função expand() do pacote tidyr. Essa função busca expandir uma tabela, de forma que ela inclua todas as possíveis combinações de certos valores. Em maiores detalhes, essa função irá criar (com base nos dados que você fornecer a ela) uma nova tabela, ou um novo tibble, que irá incluir todas as combinações únicas e possíveis dos valores que você definiu. Portanto, se eu fornecer a tabela vendas à função, e pedir a ela que encontre todas as combinações possíveis entre os valores contidos nas colunas datas e nomes, esse será o resultado:
expand(vendas, datas, nome)## # A tibble: 12 × 2
## datas nome
## <date> <chr>
## 1 2020-09-01 Ana
## 2 2020-09-01 Joao
## 3 2020-09-01 Julia
## 4 2020-09-05 Ana
## 5 2020-09-05 Joao
## 6 2020-09-05 Julia
## 7 2020-09-07 Ana
## 8 2020-09-07 Joao
## 9 2020-09-07 Julia
## 10 2020-09-10 Ana
## 11 2020-09-10 Joao
## 12 2020-09-10 JuliaPortanto, expand() irá criar uma nova tabela, contendo todas as possíveis combinações entre os valores das colunas datas e nomes da tabela vendas. Incluindo aquelas combinações que não aparecem na tabela inicial. Por exemplo, as combinações (2020-09-01, Julia), ou (2020-09-05, Joao) e (2020-09-10, Joao) não estão presentes na tabela vendas, e mesmo assim foram introduzidas no resultado de expand().
Porém, expand() não definiu novas combinações com as datas que estão faltando na tabela vendas (por exemplo, os dias 02, 03, 04 e 08 de setembro de 2020). Ou seja, em nenhum momento expand() irá adicionar algum dado à sua tabela, seja antes ou depois de encontrar todas as combinações únicas. Em outras palavras, expand() irá sempre encontrar todas as combinações possíveis, se baseando nos valores que já se encontram nas variáveis que você forneceu a ela. Por isso, mesmo que a combinação (2020-09-01, Julia) não esteja definida na tabela vendas, ela é uma combinação possível, pois os valores 2020-09-01 e Julia estão presentes na tabela vendas.
Vale destacar, que você pode combinar as variáveis de sua tabela, com vetores externos. Por exemplo, eu posso utilizar seq.Date() para gerar todas as datas que estão entre o dia 01 e 10 de setembro de 2020. No exemplo abaixo, perceba que expand() pega cada um dos 3 nomes únicos definidos na coluna nome de vendas, e combina eles com cada uma das 10 datas guardadas no vetor vec_d, gerando assim, uma nova tabela com 30 linhas (\(3\) nomes \(\times 10\) datas \(= 30\) combinações).
## # A tibble: 30 × 2
## nome vec_d
## <chr> <date>
## 1 Ana 2020-09-01
## 2 Ana 2020-09-02
## 3 Ana 2020-09-03
## 4 Ana 2020-09-04
## 5 Ana 2020-09-05
## 6 Ana 2020-09-06
## 7 Ana 2020-09-07
## 8 Ana 2020-09-08
## 9 Ana 2020-09-09
## 10 Ana 2020-09-10
## # ℹ 20 more rowsAlém disso, a função expand() conta com uma função auxiliar útil (nesting()), que restringe quais combinações serão válidas para expand(). Ao incluir variáveis dentro da função nesting(), você está dizendo à expand(), que encontre apenas as combinações únicas (entre os valores dessas variáveis) que já estão presentes em sua base. Ou seja, se eu colocar as colunas datas e nome dentro de nesting(), a função expand() irá basicamente repetir a tabela vendas. Pois cada uma das 4 linhas (ou 4 combinações entre datas e nome), aparecem uma única vez nessa tabela.
## # A tibble: 4 × 2
## datas nome
## <date> <chr>
## 1 2020-09-01 Ana
## 2 2020-09-05 Julia
## 3 2020-09-07 Joao
## 4 2020-09-10 JuliaDessa maneira, o uso de nesting() acima, é análogo ao uso da função unique() que vêm dos pacotes básicos do R. Logo, poderíamos atingir exatamente o mesmo resultado, utilizando qualquer uma das duas funções. Podemos por exemplo, adicionarmos uma quinta linha à tabela vendas, que repete os valores contidos na quarta linha da tabela. Perceba abaixo, que ao utilizarmos unique() ou nesting(), em ambos os casos, essa quinta linha repetida desaparece. Pois ambas as funções buscam encontrar todas as combinações únicas que aparecem ao longo da tabela vendas.
vendas[5, ] <- data.frame(as.Date("2020-09-10"), "Julia", 739.29)
vendas## # A tibble: 5 × 3
## datas nome valor
## <date> <chr> <dbl>
## 1 2020-09-01 Ana 406.
## 2 2020-09-05 Julia 528.
## 3 2020-09-07 Joao 375.
## 4 2020-09-10 Julia 739.
## 5 2020-09-10 Julia 739.
# Estou aplicando unique() sobre a
# primeira e segunda coluna de vendas
unique(vendas[ , 1:2]) ## # A tibble: 4 × 2
## datas nome
## <date> <chr>
## 1 2020-09-01 Ana
## 2 2020-09-05 Julia
## 3 2020-09-07 Joao
## 4 2020-09-10 Julia
# O mesmo resultado pode ser
# atingido com o uso de nesting() em expand()
expand(vendas, nesting(datas, nome))## # A tibble: 4 × 2
## datas nome
## <date> <chr>
## 1 2020-09-01 Ana
## 2 2020-09-05 Julia
## 3 2020-09-07 Joao
## 4 2020-09-10 JuliaVale destacar que você pode combinar o comportamento restrito e irrestrito de expand(). Ou seja, você pode restringir as combinações com o uso de nesting() para algumas variáveis, enquanto outras permanecem de fora dessa função, permitindo uma gama maior de combinações. No exemplo abaixo, expand() vai encontrar primeiro, cada combinação única entre nome e valor que está presente na tabela vendas, em seguida, a função irá encontrar todas as combinações possíveis entre as combinações anteriores (entre nome e valor) e todas as datas descritas na base.
Em outras palavras, nós podemos encontrar no resultado abaixo, uma combinação como (2020-09-01, Julia, 528.). Pois a combinação (Julia, 528.) existe nas colunas nome e valor da tabela vendas, e como deixamos a coluna datas de fora de nesting(), expand() irá combinar (Julia, 528.) com toda e qualquer data disponível na tabela vendas.
Porém, nós não podemos encontrar no resultado abaixo, uma combinação como (2020-09-01, Ana, 739.). Pois a única combinação entre as colunas nome e valor, presente na tabela vendas, que possui o valor 739 na coluna valor, é a linha que contém a combinação (Julia, 739.). Logo, se não há nas colunas nome e valor alguma combinação entre Ana e o valor 739., expand() não irá combinar esses valores com todas as datas disponíveis na base. Pois as combinações entre as colunas nome e valor estão sendo restringidas por nesting().
## # A tibble: 16 × 3
## datas nome valor
## <date> <chr> <dbl>
## 1 2020-09-01 Ana 406.
## 2 2020-09-01 Joao 375.
## 3 2020-09-01 Julia 528.
## 4 2020-09-01 Julia 739.
## 5 2020-09-05 Ana 406.
## 6 2020-09-05 Joao 375.
## 7 2020-09-05 Julia 528.
## 8 2020-09-05 Julia 739.
## 9 2020-09-07 Ana 406.
## 10 2020-09-07 Joao 375.
## 11 2020-09-07 Julia 528.
## 12 2020-09-07 Julia 739.
## 13 2020-09-10 Ana 406.
## 14 2020-09-10 Joao 375.
## 15 2020-09-10 Julia 528.
## 16 2020-09-10 Julia 739.7.4.2 A metodologia por detrás do processo
Apesar de próximo, a função expand() não é suficiente para produzirmos o resultado que desejamos. Lembre-se que nós temos a tabela abaixo, e que desejamos completá-la com os dados referentes aos dias 02, 03, 04, 06, 08 e 09 de setembro de 2020, que estão no momento faltando nessa tabela.
vendas## # A tibble: 4 × 3
## datas nome valor
## <date> <chr> <dbl>
## 1 2020-09-01 Ana 406.
## 2 2020-09-05 Julia 528.
## 3 2020-09-07 Joao 375.
## 4 2020-09-10 Julia 739.Primeiro, precisamos encontrar todos valores possíveis da variável que está incompleta na tabela vendas. Ou seja, queremos encontrar todas as datas possíveis entre os dias 01 e 10 de setembro de 2020, pois esses dias são os limites da tabela. Dito de outra forma, a tabela vendas descreve dados de vendas que ocorreram do dia 01 até o dia 10 de setembro de 2020. Por isso, queremos encontrar todos os dias possíveis entre esse intervalo de tempo.
Para isso, podemos utilizar a função seq.Date() em conjunto com tibble(). Dessa forma, nos criamos uma nova tabela, que contém uma sequência de datas que vai do dia 01 até o dia 10 de setembro. O mesmo resultado, poderia ser atingido, caso utilizássemos seq.Date() dentro de expand(), já que expand() cria por padrão uma nova tabela com todas as combinações possíveis dos dados que você fornece a ela.
## # A tibble: 10 × 1
## datas
## <date>
## 1 2020-09-01
## 2 2020-09-02
## 3 2020-09-03
## 4 2020-09-04
## 5 2020-09-05
## 6 2020-09-06
## 7 2020-09-07
## 8 2020-09-08
## 9 2020-09-09
## 10 2020-09-10
# O mesmo resultado poderia ser atingido com:
nova_tab <- expand(
datas = seq.Date(min(vendas$datas), max(vendas$datas), by = "day")
)Em seguida, podemos utilizar a função full_join()39 do pacote dplyr, para trazermos os dados disponíveis na tabela vendas para essa nova tabela nova_tab. Agora, nós temos uma nova tabela, que contém todos os dados que já estão definidos na tabela vendas, além dos dias que estavam faltando anteriormente, e que agora também estão definidos.
## # A tibble: 10 × 3
## datas nome valor
## <date> <chr> <dbl>
## 1 2020-09-01 Ana 406.
## 2 2020-09-02 <NA> NA
## 3 2020-09-03 <NA> NA
## 4 2020-09-04 <NA> NA
## 5 2020-09-05 Julia 528.
## 6 2020-09-06 <NA> NA
## 7 2020-09-07 Joao 375.
## 8 2020-09-08 <NA> NA
## 9 2020-09-09 <NA> NA
## 10 2020-09-10 Julia 739.O que resta agora, é preenchermos os campos com valores não-disponíveis (NA) com algum outro valor que seja mais claro, ou que indique de uma forma melhor, que não houve vendas realizadas naquele dia. Visando esse objetivo, temos a função replace_na() do pacote tidyr. Nessa função, você irá fornecer uma lista (list()) contendo os valores que vão substituir os valores NA em cada coluna de sua tabela. Essa lista precisa ser nomeada. Basta nomear cada valor substituto com o nome da coluna em que você deseja utilizar esse valor. Logo, se eu quero substituir todos os valores NA na coluna valor, por um zero, basta eu nomear esse zero com o nome dessa coluna, dentro da lista (list()) que eu forneci à replace_na().
nova_tab %>%
replace_na(
list(nome = "Não houve vendas", valor = 0)
)## # A tibble: 10 × 3
## datas nome valor
## <date> <chr> <dbl>
## 1 2020-09-01 Ana 406.
## 2 2020-09-02 Não houve vendas 0
## 3 2020-09-03 Não houve vendas 0
## 4 2020-09-04 Não houve vendas 0
## 5 2020-09-05 Julia 528.
## 6 2020-09-06 Não houve vendas 0
## 7 2020-09-07 Joao 375.
## 8 2020-09-08 Não houve vendas 0
## 9 2020-09-09 Não houve vendas 0
## 10 2020-09-10 Julia 739.
7.4.3 A função complete() como um atalho útil
A função complete() é um wrapper, ou uma função auxiliar do pacote tidyr, que engloba as funções expand(), full_join(), e replace_na(). Ou seja, a função complete() é um atalho para aplicarmos a metodologia que acabamos de descrever na seção anterior. A função possui três argumentos principais: 1) data, o nome do objeto onde a sua tabela está salva; 2) ..., a especificação das colunas a serem completadas, ou “expandidas” por complete(); 3) fill, uma lista nomeada (como a que fornecemos em replace_na()), que atribui para cada variável (ou coluna) de sua tabela, um valor a ser utilizado (ao invés de NA) para as combinações faltantes.
Vou explicar o argumento fill mais a frente, por isso, vamos nos concentrar nos outros dois. A tabela que contém os nossos dados se chama vendas, e por isso, é esse valor que devemos atribuir ao argumento data. Porém, como estamos utilizando o pipe (%>%) no exemplo abaixo, ele já está realizando esse serviço para nós. Já o segundo argumento (...), diz respeita a lista de especificações que vão definir como a função complete() deve completar cada coluna da nossa tabela.
Em outras palavras, o segundo argumento (...) é a parte da função complete() que diz respeito ao uso de expand(). Você deve, portanto, preencher este argumento, da mesma forma que você faria com a função expand(). No exemplo abaixo, o primeiro argumento (data), já está sendo definido pelo operador pipe (%>%). Perceba que eu preencho a função complete(), da mesma forma em que preenchi a função expand() na seção anterior. Perceba também, que complete() já me retorna como resultado, a tabela expandida após o uso de full_join().
## # A tibble: 10 × 3
## datas nome valor
## <date> <chr> <dbl>
## 1 2020-09-01 Ana 406.
## 2 2020-09-02 <NA> NA
## 3 2020-09-03 <NA> NA
## 4 2020-09-04 <NA> NA
## 5 2020-09-05 Julia 528.
## 6 2020-09-06 <NA> NA
## 7 2020-09-07 Joao 375.
## 8 2020-09-08 <NA> NA
## 9 2020-09-09 <NA> NA
## 10 2020-09-10 Julia 739.O último passo que resta agora, seria o uso de replace_na() para preencher os valores não-disponíveis por algum outro valor mais claro. Nós ainda podemos utilizar a função complete() para executarmos esse passo. Basta você fornecer à complete() através de seu terceiro argumento (fill), a mesma lista que você forneceria à replace_na(). Dessa forma, temos:
vendas %>%
complete(
datas = seq.Date(min(datas), max(datas), by = "day"),
fill = list(nome = "Não houve vendas", valor = 0)
)## # A tibble: 10 × 3
## datas nome valor
## <date> <chr> <dbl>
## 1 2020-09-01 Ana 406.
## 2 2020-09-02 Não houve vendas 0
## 3 2020-09-03 Não houve vendas 0
## 4 2020-09-04 Não houve vendas 0
## 5 2020-09-05 Julia 528.
## 6 2020-09-06 Não houve vendas 0
## 7 2020-09-07 Joao 375.
## 8 2020-09-08 Não houve vendas 0
## 9 2020-09-09 Não houve vendas 0
## 10 2020-09-10 Julia 739.
7.5 Preenchendo valores não-disponíveis (NA)
7.5.1 Utilizando-se de valores anteriores ou posteriores
As operações que vou mostrar a seguir, servem para preencher linhas com dados não-disponíveis (NA), com valores anteriores ou posteriores que estão disponíveis em sua tabela. Vamos começar com um exemplo simples através da tabela df, que você pode criar em seu R utilizando os comandos abaixo. Nessa tabela, temos algumas vendas anuais hipotéticas. Agora, perceba que por algum motivo, o ano em que as vendas ocorreram, só foram guardadas na primeira linha de cada ID (id). Isso é algo que devemos corrigir nessa tabela.
library(tidyverse)
v <- 2001:2004
set.seed(1)
df <- tibble(
id = rep(1:4, each = 3),
ano = NA_real_,
valor = rnorm(12, mean = 1000, sd = 560)
)
df[seq(1, 12, by = 3), "ano"] <- v
df## # A tibble: 12 × 3
## id ano valor
## <int> <dbl> <dbl>
## 1 1 2001 649.
## 2 1 NA 1103.
## 3 1 NA 532.
## 4 2 2002 1893.
## 5 2 NA 1185.
## 6 2 NA 541.
## 7 3 2003 1273.
## 8 3 NA 1413.
## 9 3 NA 1322.
## 10 4 2004 829.
## 11 4 NA 1847.
## 12 4 NA 1218.Portanto, o que queremos fazer, é completar as linhas de NA’s, com o ano correspondente a essas vendas. Pelo fato dos anos estarem separados por um número constante de linhas, ou seja, a cada 3 linhas de NA’s, temos um novo ano, podemos pensar em algumas soluções relativamente simples como a definida abaixo. Porém, a simplicidade do problema, depende dos intervalos entre cada valor, serem constantes. A partir do momento em que esses valores começarem a se dispersar em distâncias inconsistentes, uma solução como a definida abaixo, não servirá.
niveis <- unique(df$ano)
niveis <- niveis[!is.na(niveis)]
repair_vec <- df$ano
repair_vec[is.na(repair_vec)] <- rep(niveis, each = 2)
df$ano <- repair_vec
df## # A tibble: 12 × 3
## id ano valor
## <int> <dbl> <dbl>
## 1 1 2001 649.
## 2 1 2001 1103.
## 3 1 2001 532.
## 4 2 2002 1893.
## 5 2 2002 1185.
## 6 2 2002 541.
## 7 3 2003 1273.
## 8 3 2003 1413.
## 9 3 2003 1322.
## 10 4 2004 829.
## 11 4 2004 1847.
## 12 4 2004 1218.Apesar de ser um problema simples, podemos alcançar uma solução ainda mais simples, ao utilizarmos funções que são especializadas nesses problemas. Esse é o caso da função fill() do pacote tidyr, que foi criada justamente para esse propósito. Portanto, sempre que você possuir em sua tabela, uma coluna onde você deseja substituir uma sequência de NA’s pelo último (ou próximo) valor disponível, você pode utilizar essa função para tal tarefa.
A função fill() possui três argumentos: 1) data, o objeto onde a tabela com que deseja trabalhar, está salva; 2) ..., a lista de colunas em que você deseja aplicar a função; 3) .direction, define a direção que a função deve seguir na hora de preencher os valores.
## # A tibble: 12 × 3
## id ano valor
## <int> <dbl> <dbl>
## 1 1 2001 649.
## 2 1 2001 1103.
## 3 1 2001 532.
## 4 2 2002 1893.
## 5 2 2002 1185.
## 6 2 2002 541.
## 7 3 2003 1273.
## 8 3 2003 1413.
## 9 3 2003 1322.
## 10 4 2004 829.
## 11 4 2004 1847.
## 12 4 2004 1218.A função fill() trabalha a partir de uma dada direção vertical em sua tabela.
Por padrão, a função fill() irá preencher os valores indo para baixo, ou seja, partindo do topo da tabela, até a sua base. Logo, a função irá substituir qualquer NA com o último valor disponível, ou em outras palavras, com o valor disponível anterior ao NA em questão. A função lhe oferece o argumento .direction, caso você deseja alterar esse comportamento. Logo, se você deseja preencher esses valores NA’s com o próximo valor disponível em relação ao NA em questão. Isto é, preencher os valores para cima, partindo da base da tabela, e seguindo para o seu topo. Você precisa definir o argumento da seguinte maneira:
## # A tibble: 12 × 3
## id ano valor
## <int> <dbl> <dbl>
## 1 1 2001 649.
## 2 1 2002 1103.
## 3 1 2002 532.
## 4 2 2002 1893.
## 5 2 2003 1185.
## 6 2 2003 541.
## 7 3 2003 1273.
## 8 3 2004 1413.
## 9 3 2004 1322.
## 10 4 2004 829.
## 11 4 NA 1847.
## 12 4 NA 1218.
# Caso prefira não utilizar o pipe ( %>% ),
# ficaria dessa forma:
# fill(df, ano, .direction = "up")Portanto, se tivéssemos que colocar essas operações em uma representação visual, teríamos algo como a figura 7.5. Lembrando que a função usa por padrão, a direção down, logo, no primeiro caso mostrado na figura, você não precisaria definir explicitamente o argumento .direction.
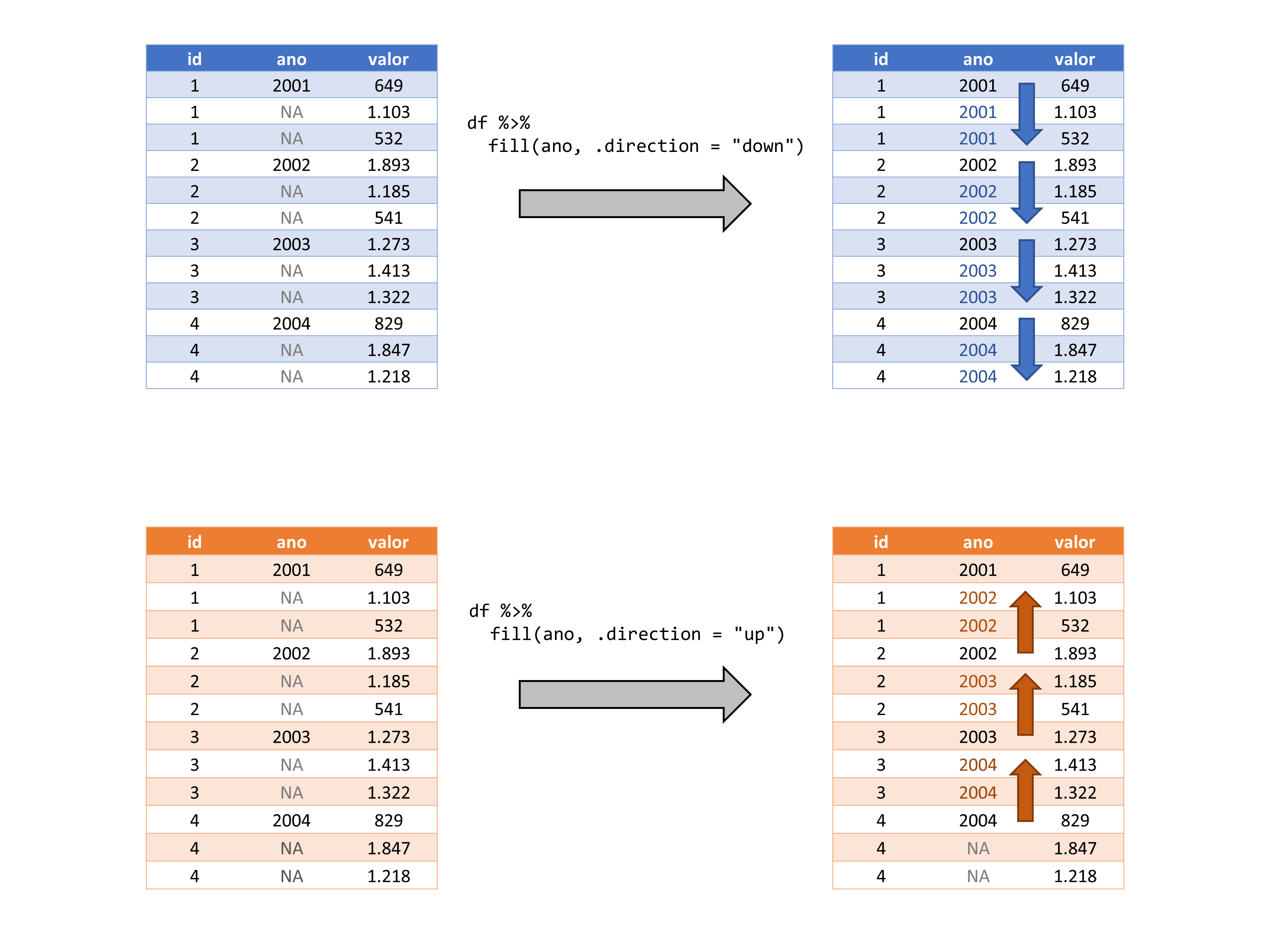
Figura 7.5: Representação do processo executado pela função fill
Apesar de serem os exemplos mais claros de aplicação, serão raras as ocasiões em que você terá esse problema posto claramente já de início em sua tabela. Com isso, eu quero dizer que serão raros os momentos em que você desde o início terá uma tabela, onde por algum motivo, os registros aparecem apenas na primeira (ou na última) linha que diz respeito aquele registro.
Usualmente, você irá utilizar a função fill() quando você já estiver realizando diversas outras transformações em sua tabela, para se chegar aonde deseja. Um exemplo claro dessa ideia, seria uma tabela onde os valores são registrados no primeiro dia de cada semana (basicamente você possui dados semanais), mas você precisa calcular uma média móvel diária. Isso significa que para calcular essa média móvel, você teria que completar os dias faltantes de cada semana, e ainda utilizar o fill() para transportar o valor do primeiro dia, para os dias restantes da semana.
Vale ressaltar, que você pode utilizar em fill(), todos os mecanismos de seleção que introduzimos em select(), para selecionar as colunas em que você deseja aplicar a função fill(). Isso também significa, que com fill() você pode preencher várias colunas ao mesmo tempo. Agora, para relembrarmos esses mecanismos, vamos criar uma tabela inicialmente vazia, que contém o total de vendas realizadas nos 6 primeiros meses de 2020, por cada funcionário de uma loja.
set.seed(2)
funcionarios <- tibble(
mes = rep(1:6, times = 4),
vendas = floor(rnorm(24, mean = 60, sd = 24)),
nome = NA_character_,
salario = NA_real_,
mes_ent = NA_real_,
ano_ent = NA_real_,
unidade = NA_character_
)
funcionarios## # A tibble: 24 × 7
## mes vendas nome salario mes_ent ano_ent unidade
## <int> <dbl> <chr> <dbl> <dbl> <dbl> <chr>
## 1 1 38 <NA> NA NA NA <NA>
## 2 2 64 <NA> NA NA NA <NA>
## 3 3 98 <NA> NA NA NA <NA>
## 4 4 32 <NA> NA NA NA <NA>
## 5 5 58 <NA> NA NA NA <NA>
## 6 6 63 <NA> NA NA NA <NA>
## 7 1 76 <NA> NA NA NA <NA>
## 8 2 54 <NA> NA NA NA <NA>
## 9 3 107 <NA> NA NA NA <NA>
## 10 4 56 <NA> NA NA NA <NA>
## # ℹ 14 more rowsEm seguida, vamos preencher as colunas vazias (nome, salario, mes_ent, …) de forma com que as informações de cada vendedor, apareçam apenas na última linha que diz respeito aquele vendedor. Como exemplo, as informações do vendedor Henrique, aparecem apenas na sexta linha da tabela, que é a última linha da tabela que se refere a ele.
valores <- list(
salario = c(1560, 2120, 1745, 1890),
nome = c("Henrique", "Ana", "João", "Milena"),
ano_ent = c(2000, 2001, 2010, 2015),
mes_ent = c(2, 10, 5, 8),
unidade = c("Afonso Pena", "Savassi", "São Paulo", "Amazonas")
)
colunas <- colnames(funcionarios)[3:7]
for(i in colunas){
funcionarios[1:4 * 6, i] <- valores[[i]]
}
# Com isso, temos o seguinte resultado:
funcionarios %>% print(n = 12)## # A tibble: 24 × 7
## mes vendas nome salario mes_ent ano_ent unidade
## <int> <dbl> <chr> <dbl> <dbl> <dbl> <chr>
## 1 1 38 <NA> NA NA NA <NA>
## 2 2 64 <NA> NA NA NA <NA>
## 3 3 98 <NA> NA NA NA <NA>
## 4 4 32 <NA> NA NA NA <NA>
## 5 5 58 <NA> NA NA NA <NA>
## 6 6 63 Henrique 1560 2 2000 Afonso Pena
## 7 1 76 <NA> NA NA NA <NA>
## 8 2 54 <NA> NA NA NA <NA>
## 9 3 107 <NA> NA NA NA <NA>
## 10 4 56 <NA> NA NA NA <NA>
## 11 5 70 <NA> NA NA NA <NA>
## 12 6 83 Ana 2120 10 2001 Savassi
## # ℹ 12 more rowsPortanto, o que precisamos é aplicar a função fill() usando .direction = "up", em cada uma dessas colunas vazias, de forma a preencher o restante das linhas com as informações de cada vendedor. Dada a natureza dessa tabela, os dois melhores mecanismos que aprendemos em select(), para selecionarmos essas colunas vazias, são: 1) usar os índices dessas colunas; 2) nos basearmos nos tipos de dados contidos em cada coluna; 3) usar um vetor externo com os nomes das colunas que desejamos.
Para utilizar o método 3 que citei acima, podemos utilizar o vetor colunas que criamos agora a pouco ao preenchermos a tabela, e já contém os nomes das colunas que desejamos. Porém, para o exemplo abaixo do método 2, você talvez se pergunte: “Se estamos aplicando a função fill() sobre todas as colunas que contém ou dados de texto (character), ou dados numéricos (numeric), nós também estamos aplicando a função sobre as colunas mes e vendas, das quais não necessitam de ajuste. O que acontece?”. Nada irá ocorrer com as colunas mes e vendas, caso elas já estejam corretamente preenchidas, portanto, podemos aplicar a função sobre elas sem medo.
# Todas as três alternativas abaixo
# geram o mesmo resultado:
funcionarios %>%
fill(
3:7,
.direction = "up"
)
funcionarios %>%
fill(
all_of(colunas),
.direction = "up"
)## # A tibble: 24 × 7
## mes vendas nome salario mes_ent ano_ent unidade
## <int> <dbl> <chr> <dbl> <dbl> <dbl> <chr>
## 1 1 38 Henrique 1560 2 2000 Afonso Pena
## 2 2 64 Henrique 1560 2 2000 Afonso Pena
## 3 3 98 Henrique 1560 2 2000 Afonso Pena
## 4 4 32 Henrique 1560 2 2000 Afonso Pena
## 5 5 58 Henrique 1560 2 2000 Afonso Pena
## 6 6 63 Henrique 1560 2 2000 Afonso Pena
## 7 1 76 Ana 2120 10 2001 Savassi
## 8 2 54 Ana 2120 10 2001 Savassi
## 9 3 107 Ana 2120 10 2001 Savassi
## 10 4 56 Ana 2120 10 2001 Savassi
## # ℹ 14 more rows
7.6 Um estudo de caso sobre médias móveis com complete() e fill()
7.6.1 A metodologia de uma média móvel no R
Uma média móvel é calculada ao aplicarmos o cálculo da média aritmética, sobre uma sequência de partes (ou subsets) de seus dados. De certa forma, esse processo se parece com uma rolagem, como se estivéssemos “rolando” o cálculo da média ao longo dos nossos dados. Veja por exemplo, o cálculo abaixo, onde utilizamos a função roll_mean() do pacote RcppRoll para calcularmos uma média móvel que possui uma janela de 3 valores.
library(RcppRoll)## [1] 2.400000 2.566667 2.100000 2.433333A janela (ou window) de uma média móvel, representa o número de observações que serão utilizadas no cálculo da média a cada “transição”, ou a cada “rolagem”. No exemplo acima, aplicamos uma média móvel com uma janela de 3 valores. Isso significa que a cada “rolagem”, são utilizados 3 valores no cálculo da média. Na primeiro rolagem, temos a média do vetor (2.7, 3.0, 1.5). Já na segunda rolagem, temos a média do vetor (3.0, 2.5, 3.2). E assim por diante. Portanto, em uma representação visual, o cálculo da média móvel aplicada por roll_mean(), é apresentado na figura 7.6.
Perceba também que o cálculo de uma média móvel implica em perda de observações. Pois no exemplo anterior, o vetor vec possui 6 valores, já o resultado de roll_mean() possui apenas 4 valores. Diversas operações estatísticas como essa, possuem o mesmo efeito. Um outro exemplo, seriam as operações de diferenciação, que são muito utilizadas em análises de séries temporais, e produzem essa mesma perda de informação. Por outro lado, no caso exposto aqui, essa perda de observações, ocorre devido ao tamanho da janela para o cálculo da média móvel.
Ou seja, pelo fato de que definimos uma janela de 3 observações para o cálculo da média móvel acima, as duas primeiras observações do vetor vec, não podem gerar a sua própria média móvel. Dito de outra forma, a função roll_mean() não pode calcular uma média nas duas primeiras rolagens sobre o vetor vec. Pois na primeira rolagem sobre o vetor, roll_mean() possui apenas uma observação (2.7). Já na segunda rolagem, roll_mean() acumula ainda duas observações (2.7, 3.0). Apenas a partir da terceira rolagem, que roll_mean() poderá calcular uma média segundo o tamanho da janela que definimos para ela, pois ela agora possui três observações (2.7, 3.0, 1.5) disponíveis para o cálculo. A partir daí, roll_mean() vai continuar rolando e calculando as médias móveis, até atingir o conjunto final de três observações do vetor vec (3.2, 1.6, 2.5).
Logo, sendo \(j\) o número de observações presentes em cada janela de cálculo de sua média móvel, e \(lvec\) sendo o número de valores presentes em seu vetor inicial, sobre o qual você irá calcular a sua média móvel. O número de médias móveis resultantes de roll_mean() (\(obs\)), será equivalente a: \(obs = lvec - (j - 1)\). Em outras palavras, o número de observações que você irá perder (\(perda\)), no cálculo de sua média móvel será equivalente a: \(perda = j - 1\).
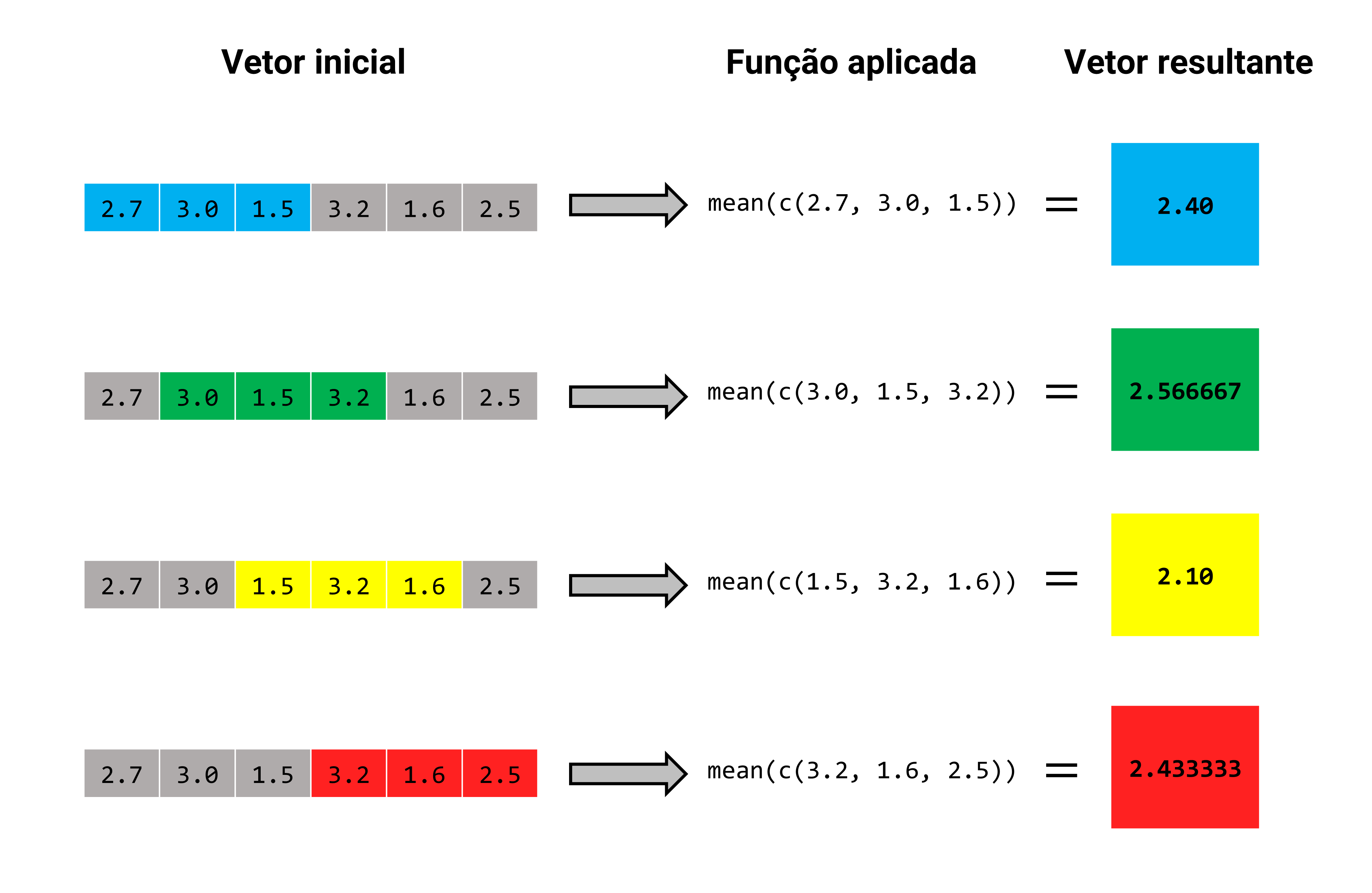
Figura 7.6: Representação do cálculo de uma média móvel
Agora, pode ser de seu desejo, contornar essa perda de observações de alguma maneira. Especialmente se você está calculando essa média móvel com base em uma coluna de sua tabela, pois sendo este o caso, provavelmente será de seu interesse, guardar essas médias calculadas em uma nova coluna dessa tabela. Entretanto, se você criar uma tabela, alocando por exemplo o vetor vec em uma coluna, e tentasse adicionar uma nova coluna contendo as médias móveis de cada ponto do vetor, o R lhe retornaria o erro abaixo. Caso você se lembre das propriedades dos data.frame’s no R, você irá entender o porquê que está motivando esse erro. Pois todas as colunas de um data.frame devem possuir obrigatoriamente o mesmo número de observações. Como nós perdemos duas das seis observações de vec, no cálculo da média móvel, o R não permite alocarmos diretamente essas médias em nossa tabela df.
df <- data.frame(x = vec)
df$media_movel <- roll_mean(df$x, n = 3)Error in `$<-.data.frame`(`*tmp*`, media_movel, value = c(2.4,
2.56666666666667, : replacement has 4 rows, data has 6.A função roll_mean() oferece duas formas de contornar esse problema: 1) definir o alinhamento da função, para preencher as observações faltantes com valores não-disponíveis (NA); 2) ou preencher essas observações faltantes com um valor pré-definido, através de seu argumento fill. Ambas formas são válidas e possivelmente são o que você deseja.
O primeiro método que citei, envolve o alinhamento da função, que você irá definir através dos sufixos r e l no nome da função. A diferença entre os dois tipos de alinhamento, decide em que parte (no início, ou no final) do vetor resultante de roll_mean(), os valores não-disponíveis (NA) serão posicionados. Se você deseja utilizar o alinhamento à direita (right - r), você deve utilizar a função roll_meanr(). Mas se você quer utilizar o alinhamento à esquerda (left - l), você deve utilizar a função roll_meanl().
Quanto ao segundo método que citei, que envolve o argumento fill, você pode utilizar o argumento align, para definir em que partes do vetor resultante, o valor que você definiu em fill será posicionado. Por exemplo, caso eu use o valor center em align, o valor definido em fill vai aparecer tanto no início quanto ao fim do vetor que resulta da função roll_mean(). Mas se eu utilizar o valor right em align, esse valor irá aparecer ao início do vetor resultante.
roll_meanr(vec, n = 3)## [1] NA NA 2.400000 2.566667 2.100000 2.433333
roll_meanl(vec, n = 3)## [1] 2.400000 2.566667 2.100000 2.433333 NA NA
roll_mean(vec, n = 3, fill = 0, align = "center")## [1] 0.000000 2.400000 2.566667 2.100000 2.433333 0.000000
roll_mean(vec, n = 3, fill = 0, align = "right")## [1] 0.000000 0.000000 2.400000 2.566667 2.100000 2.433333
roll_mean(vec, n = 3, fill = 0, align = "left")## [1] 2.400000 2.566667 2.100000 2.433333 0.000000 0.0000007.6.2 Os dados da Covid-19
Na próxima seção, busco dar um exemplo prático de como as funções complete() e fill() que vimos nas seções anteriores, podem ser utilizadas em conjunto em um problema real. Para isso, vamos utilizar parte dos dados sobre a Covid-19 (SARS-COV-2) no Brasil. Ao utilizar o código abaixo, lembre-se de renomear a primeira coluna da tabela para dia. Dessa forma, nós evitamos confusões com qualquer função que possua um argumento chamado data (funções como mutate(), select(), complete(), lm() e muitas outras possuem tal argumento).
library(tidyverse)
github <- "https://raw.githubusercontent.com/pedropark99/"
arquivo <- "Curso-R/master/Dados/covid.csv"
covid <- read_csv2(paste0(github, arquivo))
colnames(covid)[1] <- "dia"A Fundação João Pinheiro (FJP-MG) tem dado apoio técnico ao governo de Minas Gerais, no acompanhamento da pandemia de COVID-19, ao gerar estatísticas e estimações epidemiológicas para o estado. Eu fiz parte desse esforço por algum tempo, e uma demanda real que havia chegado para mim na época, consistia no cálculo de uma média móvel dos novos casos diários da doença para cada estado do Brasil. Pois era de desejo da Secretaria Estadual de Saúde, comparar a curva dessa média móvel do estado de Minas Gerais, com a de outros estados brasileiros.
Na época em que trabalhei com a base covid, ela possuía algumas barreiras, que superei com o uso de complete() e fill(). São essas barreiras, e suas resoluções que busco mostrar nessa seção, como um exemplo real de uso dessas funções. Porém, a base covid que está disponível hoje, e que você acaba de importar através dos comandos acima, é a base já corrigida e reformatada e, por isso, ela já se encontra em um formato ideal para o cálculo de uma média móvel. Portanto, antes de partirmos para a prática, vou aplicar algumas transformações, com o objetivo de “corromper” a base covid até o seu ponto inicial. Pois o foco nessa seção, se encontra na demonstração dos problemas de formatação da base, e em suas possíveis soluções.
O primeiro ponto a ser discutido, são as datas iniciais da pandemia em cada estado brasileiro. No Brasil, a pandemia de Covid-19 atingiu primeiramente o estado de São Paulo, e chegou posteriormente aos demais estados. Como podemos ver pelo resultado abaixo, a data inicial de cada estado, ao longo da base covid é difusa. Em alguns estados, os registros se iniciam a partir da data do primeiro registro de casos da doença (como os estados do Acre, Alagoas, Bahia, Amazonas e Espírito Santo). Alguns estados, registraram mais de um caso já no primeiro dia (como o Acre, que registrou três casos no dia 17 de março, e o Ceará, que reportou nove casos no dia 16 de março). Porém, outros estados (como a Paraíba) não seguem esse padrão, pois no seu primeiro dia de registro, o número de casos reportados foi igual a zero. Ou seja, a pandemia no estado da Paraíba não se iniciou oficialmente no dia 12 de março, pois não havia casos reportados até este dia.
data_inicial <- covid %>%
group_by(estado) %>%
summarise(
data_inicial = min(dia),
casos_inicial = min(casos)
)
data_inicial %>% print(n = 15)## # A tibble: 27 × 3
## estado data_inicial casos_inicial
## <chr> <date> <dbl>
## 1 AC 2020-03-17 3
## 2 AL 2020-03-08 1
## 3 AM 2020-03-13 2
## 4 AP 2020-03-20 1
## 5 BA 2020-03-06 1
## 6 CE 2020-03-16 9
## 7 DF 2020-03-07 1
## 8 ES 2020-03-05 1
## 9 GO 2020-03-12 3
## 10 MA 2020-03-20 1
## 11 MG 2020-03-08 1
## 12 MS 2020-03-14 2
## 13 MT 2020-03-20 1
## 14 PA 2020-03-18 1
## 15 PB 2020-03-12 0
## # ℹ 12 more rowsIsso não representa um grande problema, mas antes das próximas transformações, devemos iniciar os dados de cada estado, no dia de primeiro registro de casos da doença. Isto é, os dados da Paraíba, por exemplo, devem se iniciar no dia em que houve pela primeira vez, um registro de casos maior do que zero. Como a coluna casos, representa o número acumulado de casos da doença, nós podemos realizar esse “nivelamento” entre os estados, ao eliminarmos da base, todas as linhas que possuem um número acumulado de casos igual a zero. Porém, vale a pena olharmos mais atentamente sobre essas linhas antes de eliminá-las, para termos certeza de que não estamos causando mais danos ao processo.
Perceba pelo resultado abaixo, que todos as linhas em que o número acumulado de casos se iguala a zero, pertencem ao estado da Paraíba. Todas essas seis datas, são “inúteis” para o propósito da base covid, pois apresentam um cenário anterior à pandemia no estado da Paraíba. Portanto, antes de prosseguirmos, vamos eliminar essas linhas, com o uso de filter().
## # A tibble: 6 × 4
## dia estado casos mortes
## <date> <chr> <dbl> <dbl>
## 1 2020-03-12 PB 0 0
## 2 2020-03-13 PB 0 0
## 3 2020-03-14 PB 0 0
## 4 2020-03-15 PB 0 0
## 5 2020-03-16 PB 0 0
## 6 2020-03-17 PB 0 0
covid <- filter(covid, casos != 0)A base covid atualmente possui os números de casos diários acumulados de cada estado brasileiro. Mas vamos supor, que a base covid registrasse o número de casos acumulados, somente nos dias em que esse número se alterasse. Ou seja, se o número de casos acumulados da doença em uma segunda-feira qualquer do ano, era de 300, e esse número se manteve constante ao longo da semana, até que na sexta-feira, esse número subiu para 301 casos, a base covid irá registrar os números de casos acumulados apenas para as datas da segunda e da sexta dessa semana. Tal resultado pode ser atingido com os comandos abaixo. Como nós filtramos anteriormente a base, de forma a retirar as linhas com valores iguais a zero na coluna casos, temos que reconstruir o objeto data_inicial, como exposto abaixo.
data_inicial <- covid %>%
group_by(estado) %>%
summarise(
data_inicial = min(dia),
casos_inicial = min(casos)
) %>%
mutate(
dia = as.Date(data_inicial - 1),
casos = NA_real_,
mortes = NA_real_
)
covid <- covid %>%
bind_rows(
data_inicial %>% select(dia, estado, casos, mortes)
) %>%
group_by(estado) %>%
arrange(
dia,
estado,
.by_group = TRUE
) %>%
mutate(
teste = lead(casos) == casos
) %>%
filter(teste == FALSE) %>%
ungroup() %>%
select(-teste)Portanto, temos agora a tabela abaixo, onde podemos perceber que no dia 21 de março de 2020 não houve alteração no número de casos acumulados no estado do Acre. Pois esse dia (2020-03-21) não está mais presente na tabela covid. Dito de outra forma, o número de novos casos de Covid-19 que surgiram no dia 21 de março, foi igual a zero. O mesmo ocorre com os dias 25 e 27 de março no estado, que também não estão mais presentes na base.
covid## # A tibble: 3,456 × 4
## dia estado casos mortes
## <date> <chr> <dbl> <dbl>
## 1 2020-03-18 AC 3 0
## 2 2020-03-19 AC 4 0
## 3 2020-03-20 AC 7 0
## 4 2020-03-22 AC 11 0
## 5 2020-03-23 AC 17 0
## 6 2020-03-24 AC 21 0
## 7 2020-03-26 AC 23 0
## 8 2020-03-28 AC 25 0
## 9 2020-03-29 AC 34 0
## 10 2020-03-30 AC 41 0
## # ℹ 3,446 more rowsQuando trabalhei anteriormente com a base covid anteriormente, ela se encontrava inicialmente em um formato muito próximo deste. Por isso, a base necessitava de ajustes para o cálculo da média móvel. O intuito da próxima seção, é demonstrar como eu fiz esses ajustes necessários, através das funções complete() e fill().
7.6.3 Buscando soluções com complete() e fill()
Considerando que você aplicou as transformações expostas na seção anterior (Os dados da Covid-19), você está apto a aplicar os comandos apresentados nessa seção. Agora, em que sentido essa nova tabela que temos, é inapropriada para o cálculo da média móvel diária de novos casos? Porque agora faltam os registros dos dias em que não houve alteração no número acumulado de casos em cada estado. Ou seja, nós retiramos na seção anterior, justamente aquilo que queremos recuperar nessa seção. Como eu disse, o intuito dessas seções, está nos exemplos de uso das funções complete() e fill(), e não no caminho que temos que percorrer para estarmos aptos para a aplicação desses exemplos.
Portanto, o problema que possuímos agora no cálculo da média móvel sobre a base covid, é que faltam os dias onde o número de casos acumulados permaneceu constante. Isso significa que agora temos uma quebra no cálculo da média móvel. Caso o vetor vec abaixo, representasse uma parte da coluna casos da nossa base covid, se aplicássemos uma média móvel, com uma janela de 3 valores, as médias dos dias 03 e 04 não poderiam ser calculadas (ou no mínimo, estariam incorretas), tendo em vista as transformações que aplicamos na seção anterior.
Ou seja, considerando que nós eliminamos na seção anterior, todas as linhas de covid, onde o número acumulado de casos permaneceu constante em relação ao seu valor anterior; se aplicarmos a mesma transformação ao vetor vec abaixo, o valor referente ao dia 02 seria eliminado, e por isso, uma quebra ocorreria sobre o cálculo das médias móveis dos dias 03 e 04. Pois, dentre os valores dos 3 dias anteriores aos dias 03 e 04, estaria faltando o valor referente ao dia 02.
vec <- c("Dia 01" = 1, "Dia 02" = 1, "Dia 03" = 3,
"Dia 04" = 5, "Dia 05" = 7)
vec## Dia 01 Dia 02 Dia 03 Dia 04 Dia 05
## 1 1 3 5 7São por essas razões, que devemos recuperar os dias perdidos na tabela covid, mesmo que o número de casos nesses dias tenham permanecido constantes. No nosso caso, não podemos utilizar diretamente as colunas da base covid, para expandirmos a tabela, e recuperarmos as datas que foram perdidas. Pois essas datas não se encontram mais na tabela covid. Lembre-se que a função complete() irá sempre trabalhar com as observações que estão presentes em sua base, caso você não forneça algo a mais, com a qual ela possa trabalhar. Por isso, teremos que gerar na função complete(), um vetor externo à base covid, de forma a incluirmos todas as datas que faltam.
Figura 7.7: Representação das séries temporais da base covid pré e pós-transformações
Antes de prosseguir, vamos compreender exatamente qual é o estado atual da base covid. Nós eliminamos (na seção anterior) parte dos dias da base. Mais especificamente aqueles dias em que o número acumulado de casos da doença, permaneceu constante em relação a seu valor anterior. Portanto, neste momento, as séries temporais do número de casos de cada estado apresentam quebras. Em uma representação visual, essas séries se assemelham no momento às linhas em cor preta, apresentadas no gráfico da figura 7.7. São essas quebras que nos impedem de calcularmos uma média móvel desses casos.
O primeiro passo, será expandir essas séries com a função complete(). Utilizando-se de um vetor (construído pela função seq.Date()), contendo desde o dia 1 da pandemia no país (dia dos primeiros casos no estado de São Paulo, onde a pandemia se iniciou) até o último dia da base. Com isso, a função complete() irá combinar cada uma dessas datas, com cada um dos 27 estados. Após essa expansão da tabela covid, as séries de cada estado vão incluir todas as datas possíveis, incluindo aquelas que originalmente não pertenciam aquele estado. Dessa forma, as séries de cada estado, vão ser equivalentes à junção das linhas pretas, azuis e vermelhas no gráfico da figura 7.7. Formando assim novamente uma série “sólida”, ou completa.
menor_data <- min(data_inicial$data_inicial)
maior_data <- max(covid$dia)
novo_covid <- covid %>%
complete(
dia = seq.Date(menor_data, maior_data, by = "day"),
estado
) %>%
group_by(estado) %>%
arrange(dia, estado, .by_group = T) %>%
ungroup()
novo_covid## # A tibble: 4,131 × 4
## dia estado casos mortes
## <date> <chr> <dbl> <dbl>
## 1 2020-02-25 AC NA NA
## 2 2020-02-26 AC NA NA
## 3 2020-02-27 AC NA NA
## 4 2020-02-28 AC NA NA
## 5 2020-02-29 AC NA NA
## 6 2020-03-01 AC NA NA
## 7 2020-03-02 AC NA NA
## 8 2020-03-03 AC NA NA
## 9 2020-03-04 AC NA NA
## 10 2020-03-05 AC NA NA
## # ℹ 4,121 more rowsO segundo passo, será “nivelar” as séries de acordo com o período inicial de cada estado. Pois, como resultado do passo anterior, as séries de todos os estados serão iguais em comprimento (ou em número de observações). Pois as séries de todos os estados, estarão incluindo desde o dia 1 da pandemia, até o último dia da pandemia. Portanto, seguindo o gráfico da figura 7.7, no segundo passo estaremos eliminando a área vermelha de cada série, de forma que as séries de cada estado vão se equivaler à junção das linhas em preto e azul. Para isso, podemos aplicar os comandos abaixo:
novo_covid <- novo_covid %>%
right_join(
data_inicial[c("estado", "data_inicial")],
by = "estado"
)
teste <- novo_covid$dia >= novo_covid$data_inicial
novo_covid <- novo_covid[teste, ]
novo_covid## # A tibble: 3,670 × 5
## dia estado casos mortes data_inicial
## <date> <chr> <dbl> <dbl> <date>
## 1 2020-03-17 AC NA NA 2020-03-17
## 2 2020-03-18 AC 3 0 2020-03-17
## 3 2020-03-19 AC 4 0 2020-03-17
## 4 2020-03-20 AC 7 0 2020-03-17
## 5 2020-03-21 AC NA NA 2020-03-17
## 6 2020-03-22 AC 11 0 2020-03-17
## 7 2020-03-23 AC 17 0 2020-03-17
## 8 2020-03-24 AC 21 0 2020-03-17
## 9 2020-03-25 AC NA NA 2020-03-17
## 10 2020-03-26 AC 23 0 2020-03-17
## # ℹ 3,660 more rowsO terceiro passo, envolve o uso de fill() para completarmos o número de casos em cada data recuperada. Lembre-se que ao expandirmos a tabela com complete(), a função preencheu os campos das colunas casos e mortes com valores não-disponíveis (NA), na linha de cada data que não estava presente anteriormente na base covid (ou seja, as datas que foram perdidas anteriormente). Portanto, todas as linhas que possuem um valor NA nessas colunas, são as linhas que correspondem aos dias em que o número acumulado de casos se manteve constante. Como esse número se manteve constante, tudo o que precisamos fazer, é utilizar fill() para puxar os valores disponíveis anteriores para esses campos.
## # A tibble: 3,670 × 5
## dia estado casos mortes data_inicial
## <date> <chr> <dbl> <dbl> <date>
## 1 2020-03-17 AC 3 0 2020-03-17
## 2 2020-03-18 AC 3 0 2020-03-17
## 3 2020-03-19 AC 4 0 2020-03-17
## 4 2020-03-20 AC 7 0 2020-03-17
## 5 2020-03-21 AC 11 0 2020-03-17
## 6 2020-03-22 AC 11 0 2020-03-17
## 7 2020-03-23 AC 17 0 2020-03-17
## 8 2020-03-24 AC 21 0 2020-03-17
## 9 2020-03-25 AC 23 0 2020-03-17
## 10 2020-03-26 AC 23 0 2020-03-17
## # ℹ 3,660 more rowsDessa forma, temos novamente, a tabela corretamente formatada, e pronta para o cálculo de uma média móvel. O número acumulado de casos certamente tende a aumentar com o tempo, mas será que a variação desse número, segue o mesmo padrão? Para calcularmos essa variação, podemos utilizar a função lag() para utilizarmos o valor da linha anterior de uma coluna. Com isso, podemos subtrair o valor da linha anterior, sobre o valor da linha atual, tirando assim, a diferença ou a variação entre elas. Em seguida, basta aplicarmos a função roll_meanr() sobre esta variação, para adquirirmos uma média móvel do número de novos casos diários.
library(RcppRoll)
novo_covid <- novo_covid %>%
group_by(estado) %>%
mutate(
novos_casos = casos - lag(casos),
media_casos = roll_meanr(novos_casos, n = 5)
)
novo_covid## # A tibble: 3,670 × 7
## # Groups: estado [27]
## dia estado casos mortes data_inicial novos_casos media_casos
## <date> <chr> <dbl> <dbl> <date> <dbl> <dbl>
## 1 2020-03-17 AC 3 0 2020-03-17 NA NA
## 2 2020-03-18 AC 3 0 2020-03-17 0 NA
## 3 2020-03-19 AC 4 0 2020-03-17 1 NA
## 4 2020-03-20 AC 7 0 2020-03-17 3 NA
## 5 2020-03-21 AC 11 0 2020-03-17 4 NA
## 6 2020-03-22 AC 11 0 2020-03-17 0 1.6
## 7 2020-03-23 AC 17 0 2020-03-17 6 2.8
## 8 2020-03-24 AC 21 0 2020-03-17 4 3.4
## 9 2020-03-25 AC 23 0 2020-03-17 2 3.2
## 10 2020-03-26 AC 23 0 2020-03-17 0 2.4
## # ℹ 3,660 more rows
t <- "Média móvel de 5 dias para os novos casos de Covid-19 nos estados
da região Sudeste"
novo_covid %>%
filter(estado %in% c("SP", "MG", "RJ", "ES")) %>%
ggplot() +
geom_line(
aes(x = dia, y = log(media_casos), color = estado),
size = 1
) +
theme(
legend.position = "bottom",
axis.title.y = element_blank(),
plot.title = element_text(face = "bold")
) +
labs(
title = t,
subtitle = "Escala logarítmica",
x = "Tempo",
color = "Unidade da Federação"
)## Warning: Removed 20 rows containing missing values (`geom_line()`).Exercícios
Questão 7.1. Os itens desta questão vão utilizar a tabela world_bank_pop. Essa tabela advém do pacote tidyr, logo, caso você tenha chamado com sucesso por esse pacote através do comando library() você já possui acesso a essa tabela. A tabela world_bank_pop contém uma série histórica de vários dados populacionais para cada país descrito na base.
world_bank_pop## # A tibble: 1,064 × 20
## country indicator `2000` `2001` `2002` `2003` `2004` `2005` `2006`
## <chr> <chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 ABW SP.URB.T… 4.16e4 4.20e+4 4.22e+4 4.23e+4 4.23e+4 4.24e+4 4.26e+4
## 2 ABW SP.URB.G… 1.66e0 9.56e-1 4.01e-1 1.97e-1 9.46e-2 1.94e-1 3.67e-1
## 3 ABW SP.POP.T… 8.91e4 9.07e+4 9.18e+4 9.27e+4 9.35e+4 9.45e+4 9.56e+4
## 4 ABW SP.POP.G… 2.54e0 1.77e+0 1.19e+0 9.97e-1 9.01e-1 1.00e+0 1.18e+0
## 5 AFE SP.URB.T… 1.16e8 1.20e+8 1.24e+8 1.29e+8 1.34e+8 1.39e+8 1.44e+8
## 6 AFE SP.URB.G… 3.60e0 3.66e+0 3.72e+0 3.71e+0 3.74e+0 3.81e+0 3.81e+0
## 7 AFE SP.POP.T… 4.02e8 4.12e+8 4.23e+8 4.34e+8 4.45e+8 4.57e+8 4.70e+8
## 8 AFE SP.POP.G… 2.58e0 2.59e+0 2.61e+0 2.62e+0 2.64e+0 2.67e+0 2.70e+0
## 9 AFG SP.URB.T… 4.31e6 4.36e+6 4.67e+6 5.06e+6 5.30e+6 5.54e+6 5.83e+6
## 10 AFG SP.URB.G… 1.86e0 1.15e+0 6.86e+0 7.95e+0 4.59e+0 4.47e+0 5.03e+0
## # ℹ 1,054 more rows
## # ℹ 11 more variables: `2007` <dbl>, `2008` <dbl>, `2009` <dbl>,
## # `2010` <dbl>, `2011` <dbl>, `2012` <dbl>, `2013` <dbl>, `2014` <dbl>,
## # `2015` <dbl>, `2016` <dbl>, `2017` <dbl>7.1.A) A tabela world_bank_pop não se encontra em um formato tidy. Indique qual (ou quais) dos pressupostos que definem o formato tidy data é (ou são) violado por essa tabela e, explique o porquê disso.
7.1.B) Repare que para além das colunas country e indicator, temos os dados populacionais espalhados ao longo de diversas colunas, onde cada coluna representa o valor dessa série histórica para um determinado ano. Utilize os conhecimentos desse capítulo para reunir essas várias colunas (que se referem a anos específicos da série) de modo que a base fique mais próxima de um formato tidy data.
7.1.C) Filtre todas as linhas da tabela que descrevem a população total de cada país (isto é, as linhas em que o valor na coluna indicator é igual ao código "SP.POP.TOTL"), em seguida, tente calcular a variação da população total entre cada ano da série, para todos os países.